4 ggplot
4.1 Introducción
En esta sección vamos a explorar la utilidad de R para la visualización de contenido demográfico. En Riffe y otros (2021) se publicó una colección de artículos sobre ejemplos de “buena” visualización en este tema. Allí reseña un poco de la historia (ver los gráficos de Perozzo ) y se concluye con algunas recomendaciones a la hora de pensar cómo mostrar lo que queremos comunicar. Es interesante la distinción entre gráfico exploratorio y gráfico explicativo.
4.2 ggplot
El paquete ggplot del entorno tidyverse tiene sus fundamentos en la gramática de gráficos (Wilkinson, 2005), entender que todo plot es una composición de elementos, más allá de su especificidad (dispersión de putnos, línea, torta, etc.). La idea es “generalizar” su construcción tal que un paquete no sea una colección de casos especiales, sino una forma particular de combinar elementos (capas). Ya lleva 10 años!
Todo gráfico esta compuesto, jearárquicamente, por:
- Datos: lo que contiene (en un formato determinado) aquello que queremos visualizar. Se utilza el argumento
data.- Capas
- Estética determinada: ¿quién es “x”?, ¿quién es “y”?, tamaño (
size), color (color) y forma (shape), entre otros. Para esto se utiliza la funciónaes(de aestethics en ingles), y permite mapear las columnas de la data a cada argumento. - Expresión geométrica de los datos. Las más comunes son:
- Puntos (
geom_point)
- Líneas (
geom_line)
- Barras (
geom_bar)
- Puntos (
- Estética determinada: ¿quién es “x”?, ¿quién es “y”?, tamaño (
- Transformación estadística (ajustar una regresión lineal, segmentar por percentiles, frecuencia de barra, etc.)
- Capas
- Sistema de coordenas y Escalas: las medidas relativas con las cuales interpretar los datos y su transformación visual. Personalización de ejes y leyenda, cambiar a escala logarítmica, asignar una escala de colores, etc.
Vamos a ver la distribución por edad de la EDER con ggplot. Para eso, primero vamos a calcular cuántas personas hay por edad. Recordemos que hay un código 9999 para valores desconocidos de edad
eder_dist <- eder_personas %>%
filter(p3b <= 103)|>
group_by(p3b) |>
summarise(n = n())
# genero un primer gráfico con los tres elementos básicos:
ggplot(data = eder_dist, # data
aes(x = p3b, y = n)) + # estética
geom_line() # geometría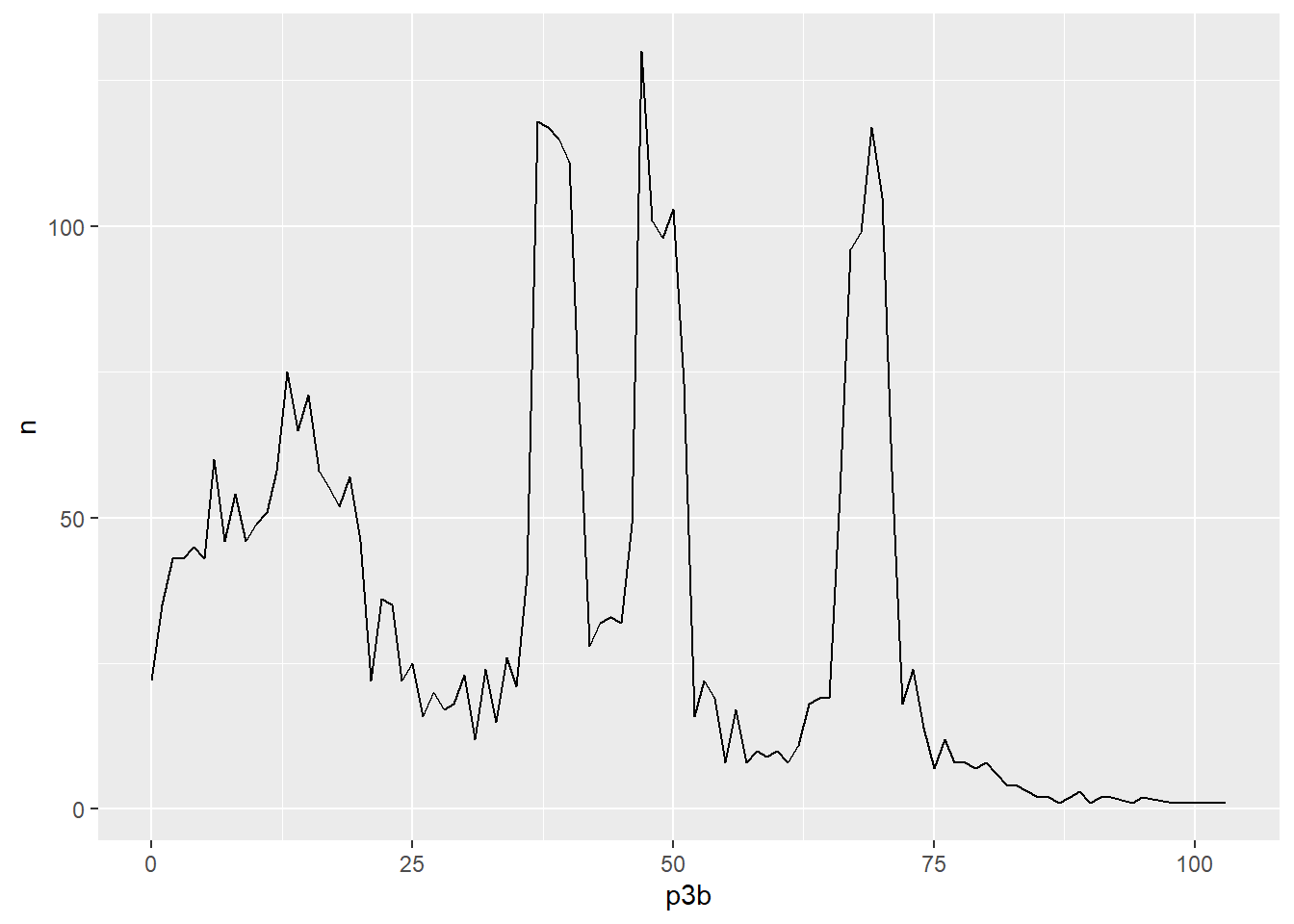
Lo bueno de %>% es que puedo verbalizar también aquí!
ggplot utiliza + en vez de %>%. No es algo de lo que esté orgulloso su creador, pero tiene sus razones.
Podemos separar mujeres y varones, distinguiéndolas por color (estética). Y vamos a sacar a la gente sin información de sexo.
eder_dist_s <- eder_personas %>%
filter(p3b <= 103,
p2 %in% 1:2)|>
group_by(p2, p3b) |> # Agrupo también por sexo
summarise(n = n())
# genero un primer gráfico con los tres elementos básicos:
eder_dist_s |>
ggplot(aes(x = p3b, y = n, color = factor(p2))) + # estética
geom_line() 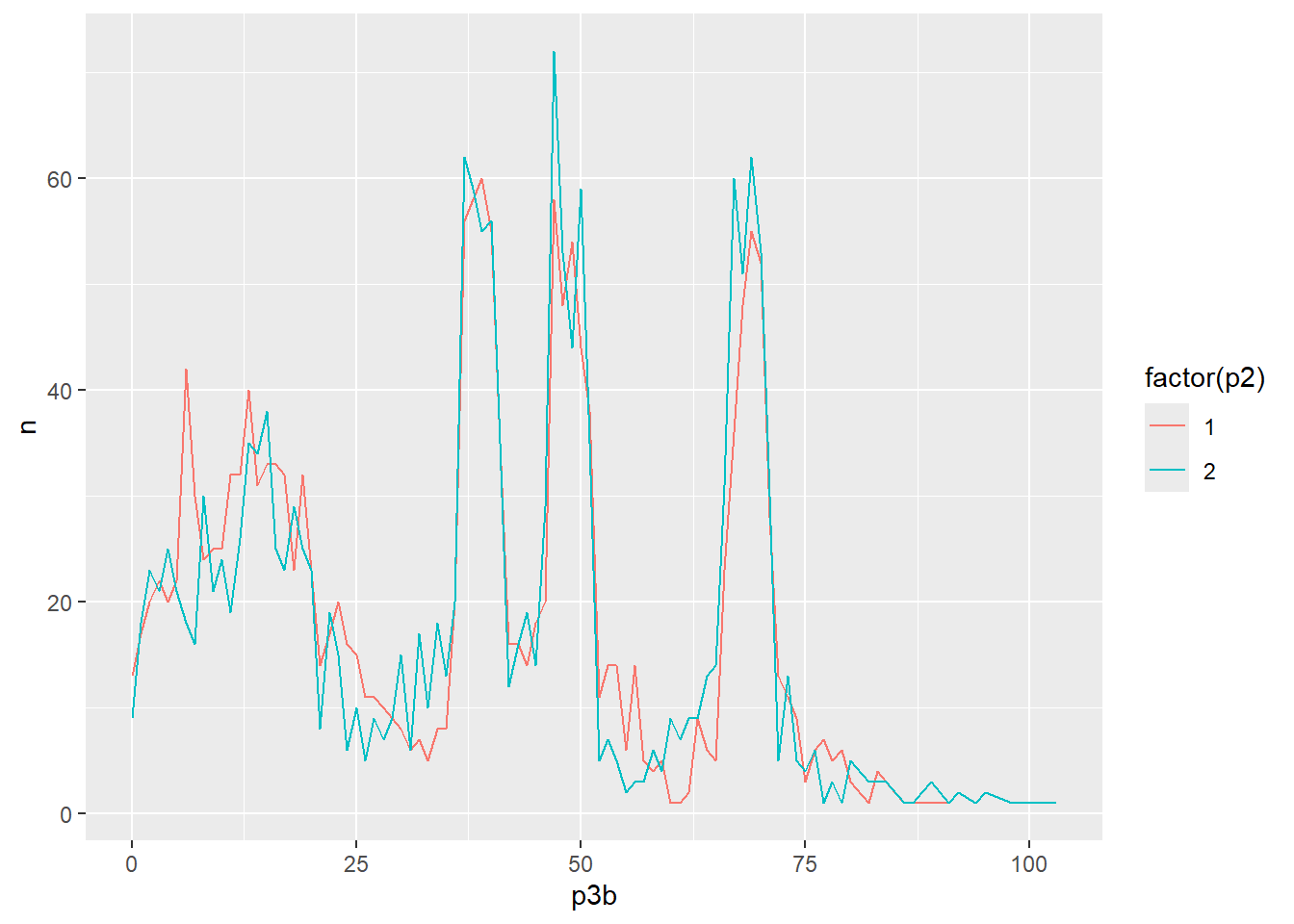
Podemos adicionar otra capa geométrica de puntos distinguiendo por forma (atributo estético adicional). Ya estamos en 4 dimensiones.
g_eder_sexo <- eder_dist_s |>
ggplot(aes(x = p3b, y = n, color = factor(p2))) + # estética
geom_line() +
geom_point(aes(shape = factor(p2)))Acabamos de crear un objeto: ¿de qué tipo es?.
Lo que se encuentra dentro de ggplot() vale para todas las capas, excepto se “pise”. Por ejemplo, da el mismo plot las siguientes dos sentencias:
data %>% ggplot(aes(x= EDAD, y = MUJER) + geom_line()
que…
data %>% ggplot() + geom_line(aes(x= EDAD, y = MUJER))
Agreguemos un título, un subtítulo, modifiquemos el tamaño del punto, la posición y título de la leyenda, y especifiquemos las marcas en el eje x:
g_eder_sexo <- g_eder_sexo + # partimos del objeto anterior
labs(title = "Población por edad y sexo. EDER 2019",
subtitle = "CABA",
caption = "Fuente: IDECBA",
x = "Edad", y = "n") +
theme(legend.position = "bottom",
legend.direction = "horizontal",
legend.title = element_blank()) +
scale_x_continuous(breaks = seq(0,100,10)) # ups!
g_eder_sexo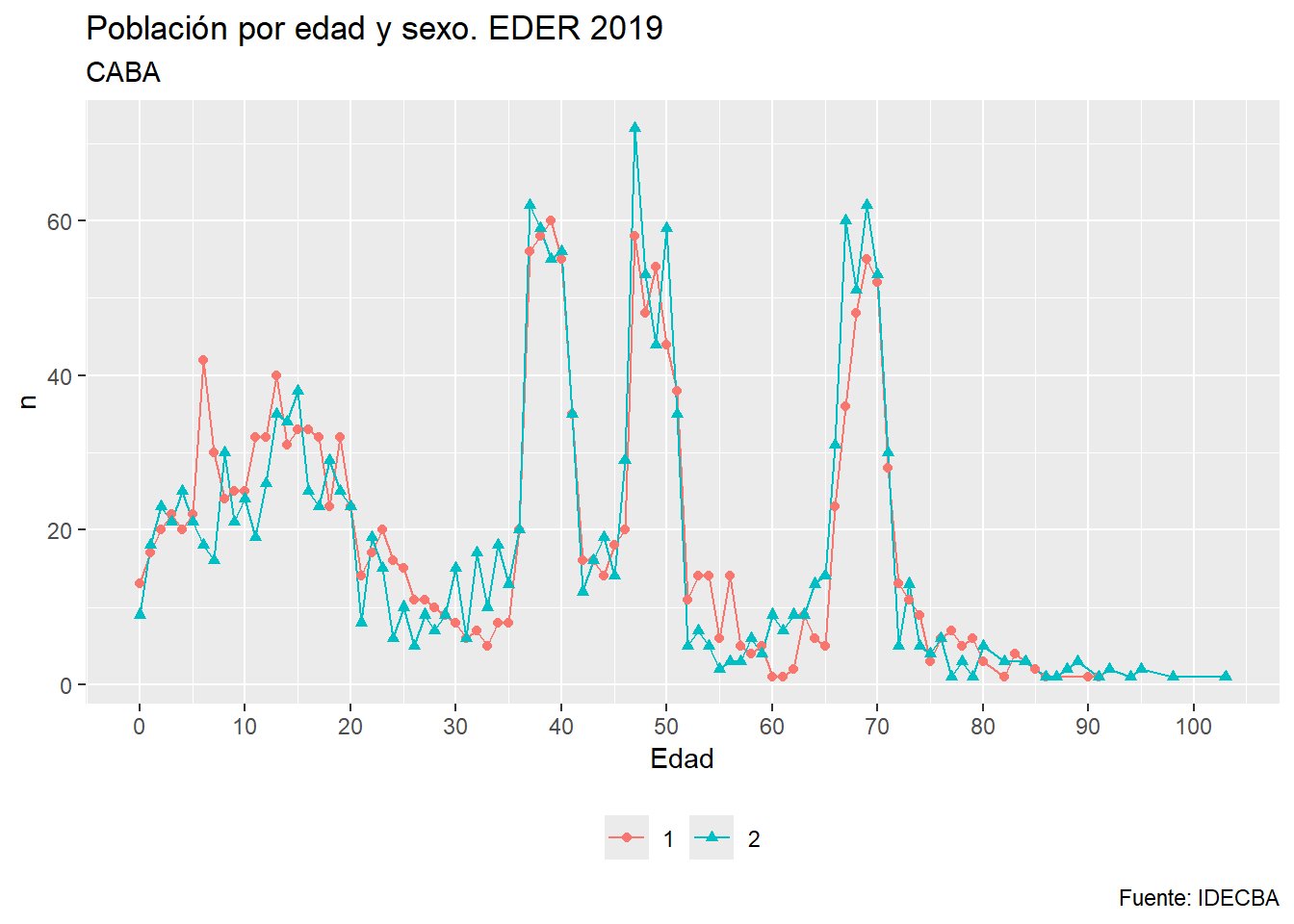
Podemos definir una paleta de colores, cambiar la temática de fondo por una más simple y suavizar las series con algún modelo. Adicionalmente mediante ggsave podemos guardar el objeto como imágen en la extensión que se desee.
mi_paleta <- c("#000000", "#E69F00", "#56B4E9") # sistema Hexadecimal de colores
g_eder_sexo <- g_eder_sexo +
scale_colour_manual(values = mi_paleta) +
theme_bw() +
geom_smooth(method = 'loess', span=.5) # podés jugar con span (0,1)
ggsave(filename = "g_eder_sexo.pdf", plot = g_eder_sexo, dpi = 150)## Saving 7 x 5 in image
## `geom_smooth()` using formula = 'y ~ x'## `geom_smooth()` using formula = 'y ~ x'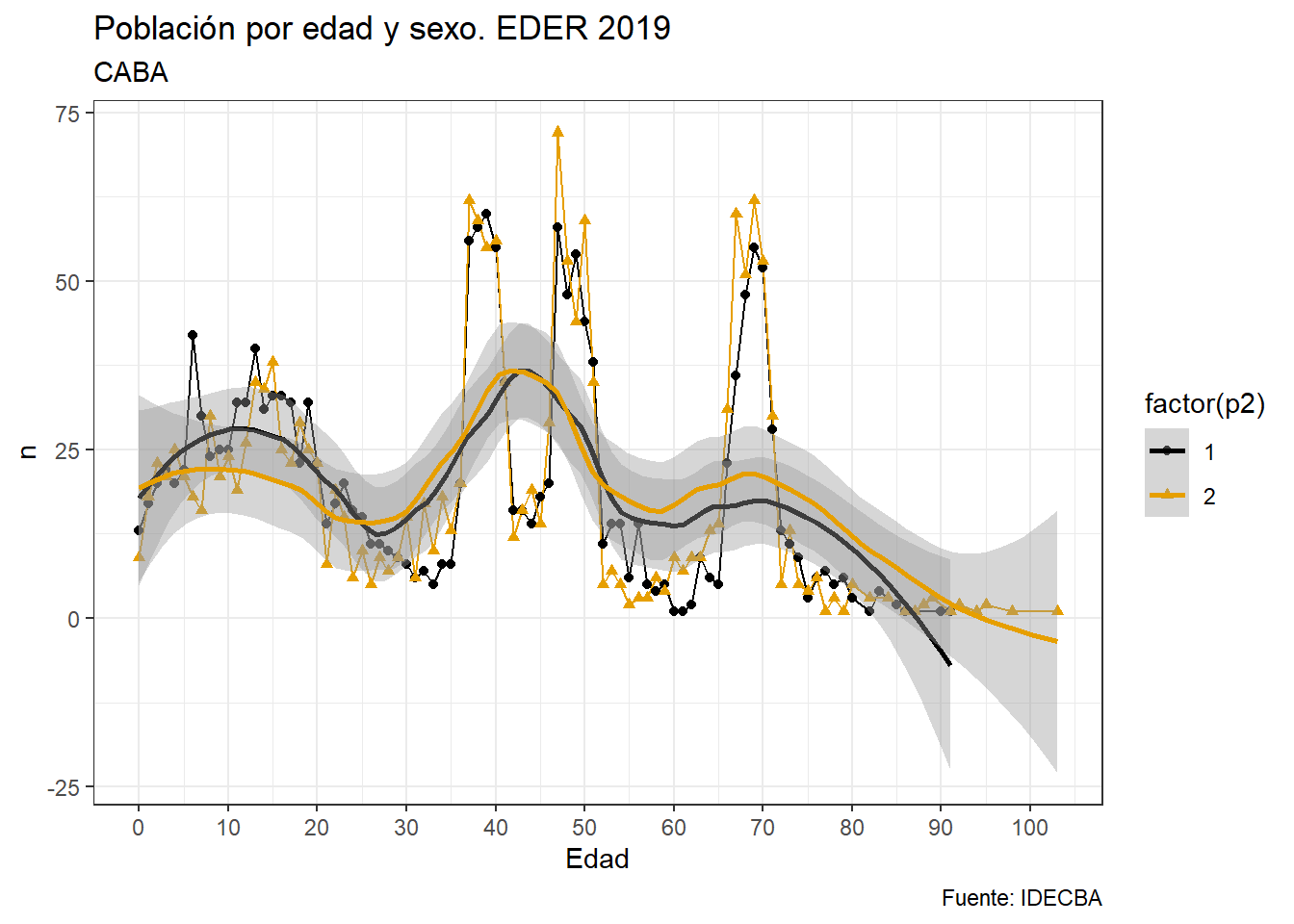
Podés encontrar más colores y paletas prediseñadas, temáticas predefinidas o incluidas en paquetes como ggthemes, y formas de suavizar una serie continua (aquí utilizamos loess, por defecto, pero puede ser lm, de linear model).
También podemos mostrar, por ejemplo, la distribución por máximo nivel educativo (una variable de tipo factor). Una buena idea: siempre tratar con tidy data!
Lo que vamos a hacer ahora, y solo a fines de entender cómo se puede armar, es llegar a un data frame donde tengamos el sexo en las columnas, y el nivel educativo en las filas.
Para esto utlizaremos las funciones pivot (antiguamaente llamadas gather y spread, por si lo ves en la web). Leamos la hoja de ayuda (cheat sheet) de tidyr juntos, otro paquete del entorno tidyverse.
eder_ned_sexo <- eder_personas %>%
filter(p2 %in% 1:2, # Elimino los que no tienen asignado el sexo y el nivel educativo
e_nivel != 9) |>
mutate(sexo = case_when(p2 == 1 ~ "Varon", # Recodifico
p2 == 2 ~ "Mujer"),
e_nivel_desc = case_when(e_nivel == 0 ~ "No corresponde/escuelas especiales",
e_nivel == 1 ~ "Inicial",
e_nivel == 2 ~ "Primario incompleto",
e_nivel == 3 ~ "Primario completo",
e_nivel == 4 ~ "Secundario incompleto",
e_nivel == 5 ~ "Secundario completo",
e_nivel == 6 ~ "Superior / Universitario incompleto",
e_nivel == 7 ~ "Superior / Universitario completo y mas",
e_nivel == 8 ~ "Sin instruccion")) |>
select(sexo, e_nivel_desc) |>
group_by(sexo, e_nivel_desc) |>
summarise(n = n()) |>
pivot_wider(names_from = sexo, values_from = n)%>%
mutate(e_nivel_desc = factor(e_nivel_desc,
levels = c("No corresponde/escuelas especiales",
"Inicial",
"Primario incompleto",
"Primario completo",
"Secundario incompleto",
"Secundario completo",
"Superior / Universitario incompleto",
"Superior / Universitario completo y mas",
"Sin instruccion"))) |>
arrange(e_nivel_desc)Es el momento de pasar a una geometría de barras. Al momento de trabajar con barras, la estética fill equivale al atributo color de líneas o puntos. Podemos incluir el condicional de signo dentro de la definición de ejes (o de manera previa mediante mutate, como prefieras):
dist_ned_muj <- eder_ned_sexo |>
ggplot(aes(x = e_nivel_desc, y = Mujer, fill = e_nivel_desc))+
geom_col()
dist_ned_muj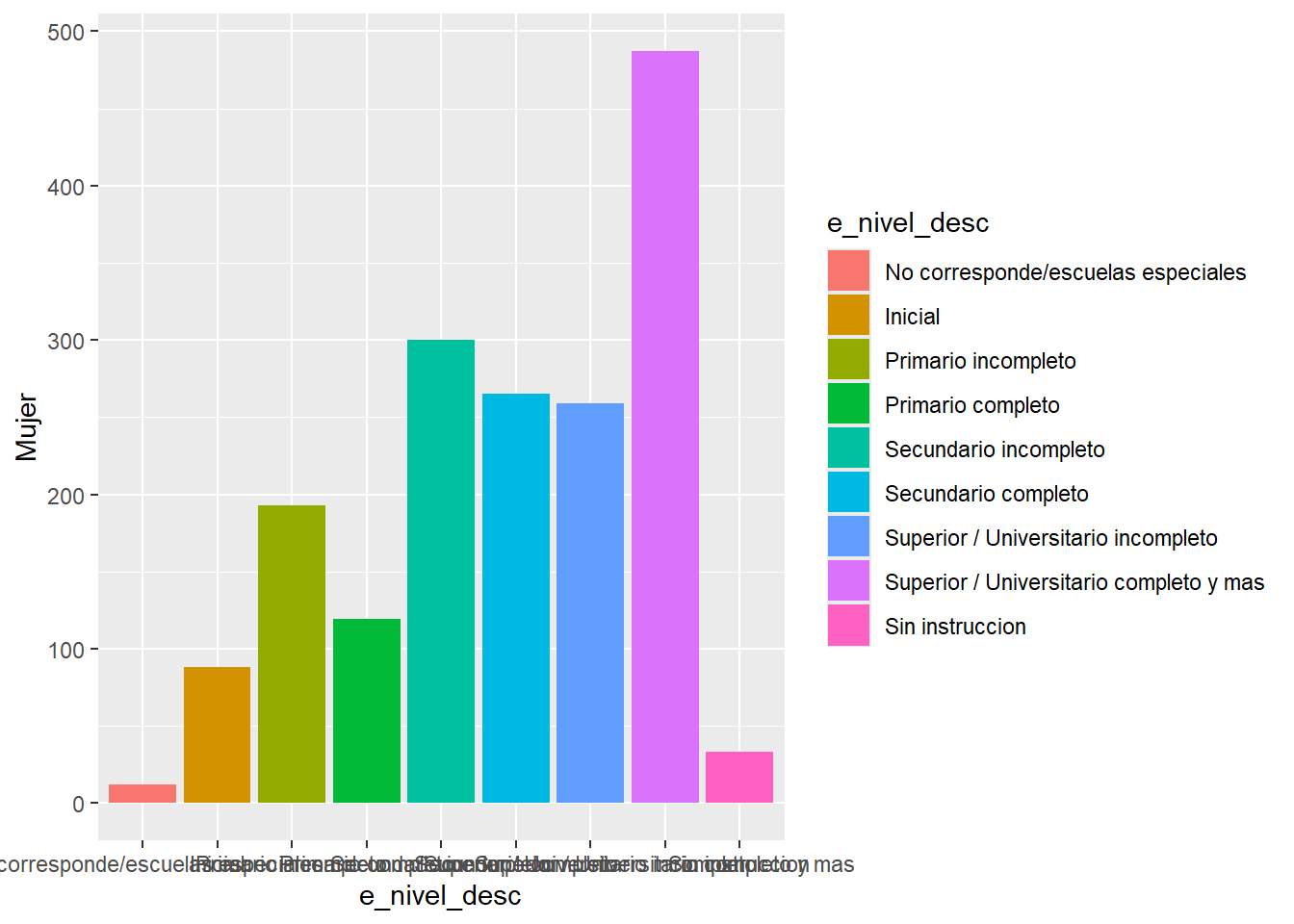
Ups! Habría que darlo vuelta (flip) para que se vea mejor. Podemos hacerle unos cambios adicionales de paso: incluir titulo, subtítulo y fuente, cambiar las etiquetas de ambos ejes, incluir un tema distinto y cambiar el color de las barras.
dist_ned_muj <- dist_ned_muj +
coord_flip() +
scale_y_continuous(labels = abs) +
labs(y = "n",
x = "Nivel educativo",
title = "Mujeres por nivel educativo",
subtitle = "EDER CABA",
caption = "Fuente: en base a IDECBA") +
theme_bw()
dist_ned_muj 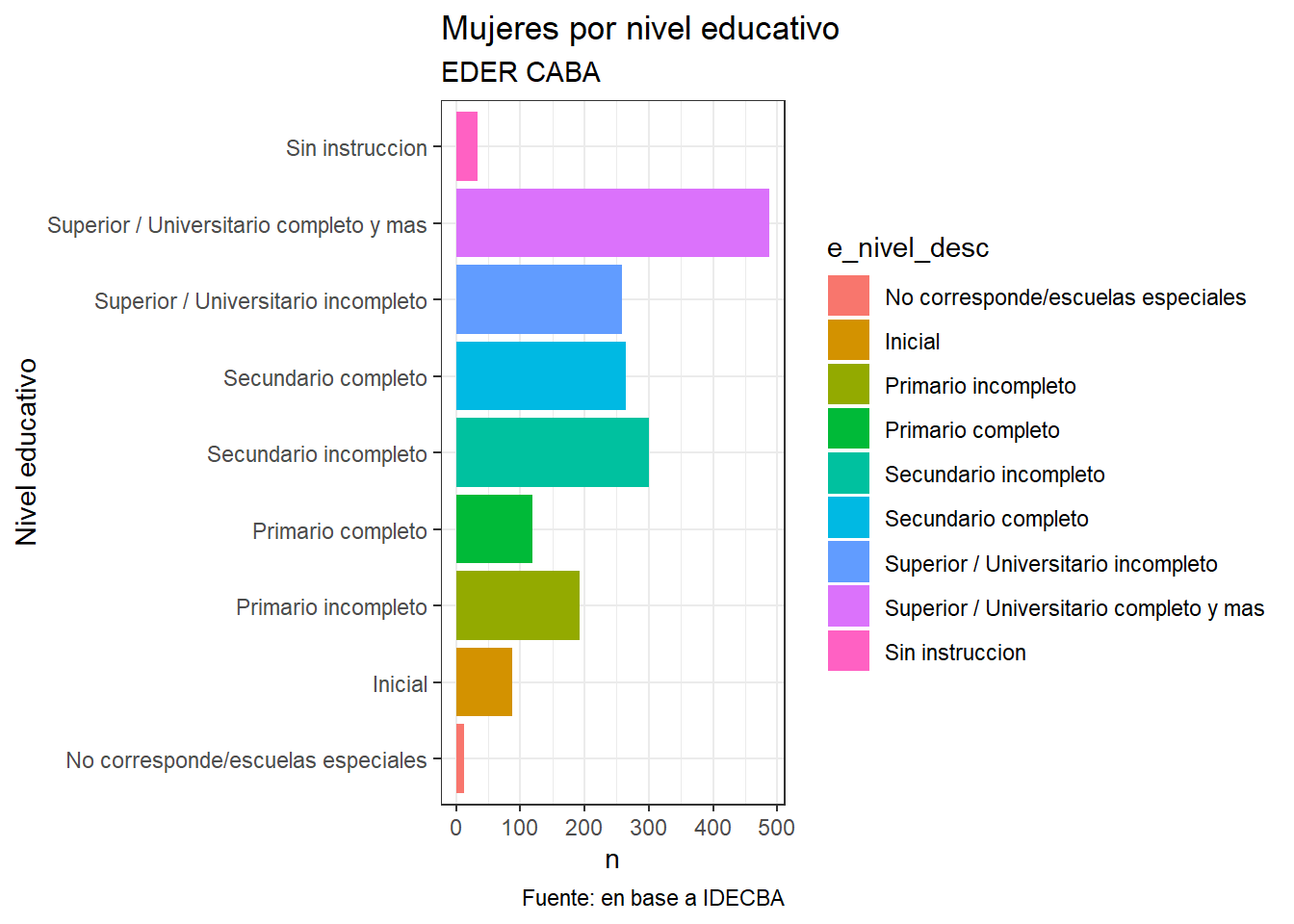
Probablemente querramos varones y mujeres. Volvamos a poner el sexo como una variable, esta vez con pivot_longer
eder_ned_sexo_long <- eder_ned_sexo |>
pivot_longer(cols = c("Mujer", "Varon"), names_to = "Sexo",values_to ="n")
eder_ned_sexo_long## # A tibble: 18 × 3
## e_nivel_desc Sexo n
## <fct> <chr> <int>
## 1 No corresponde/escuelas especiales Mujer 12
## 2 No corresponde/escuelas especiales Varon 6
## 3 Inicial Mujer 88
## 4 Inicial Varon 85
## 5 Primario incompleto Mujer 193
## 6 Primario incompleto Varon 240
## 7 Primario completo Mujer 119
## 8 Primario completo Varon 101
## 9 Secundario incompleto Mujer 300
## 10 Secundario incompleto Varon 361
## 11 Secundario completo Mujer 265
## 12 Secundario completo Varon 230
## 13 Superior / Universitario incompleto Mujer 259
## 14 Superior / Universitario incompleto Varon 297
## 15 Superior / Universitario completo y mas Mujer 487
## 16 Superior / Universitario completo y mas Varon 406
## 17 Sin instruccion Mujer 33
## 18 Sin instruccion Varon 30Y si quisiéramos ver el mismo gráfico que antes, pero separado por sexo, podemos usar las opciones de faceting. :
g_ned_sexo <- eder_ned_sexo_long %>%
ggplot(aes(x = e_nivel_desc, y = n, fill = e_nivel_desc))+
geom_col()+
coord_flip() +
scale_y_continuous(labels = abs) +
labs(y = "n",
x = "Nivel educativo",
title = "Poblacion por sexo y nivel educativo",
subtitle = "EDER CABA",
caption = "Fuente: en base a IDECBA") +
theme_dark() +
facet_grid(rows = vars(Sexo)) # o cols?
g_ned_sexo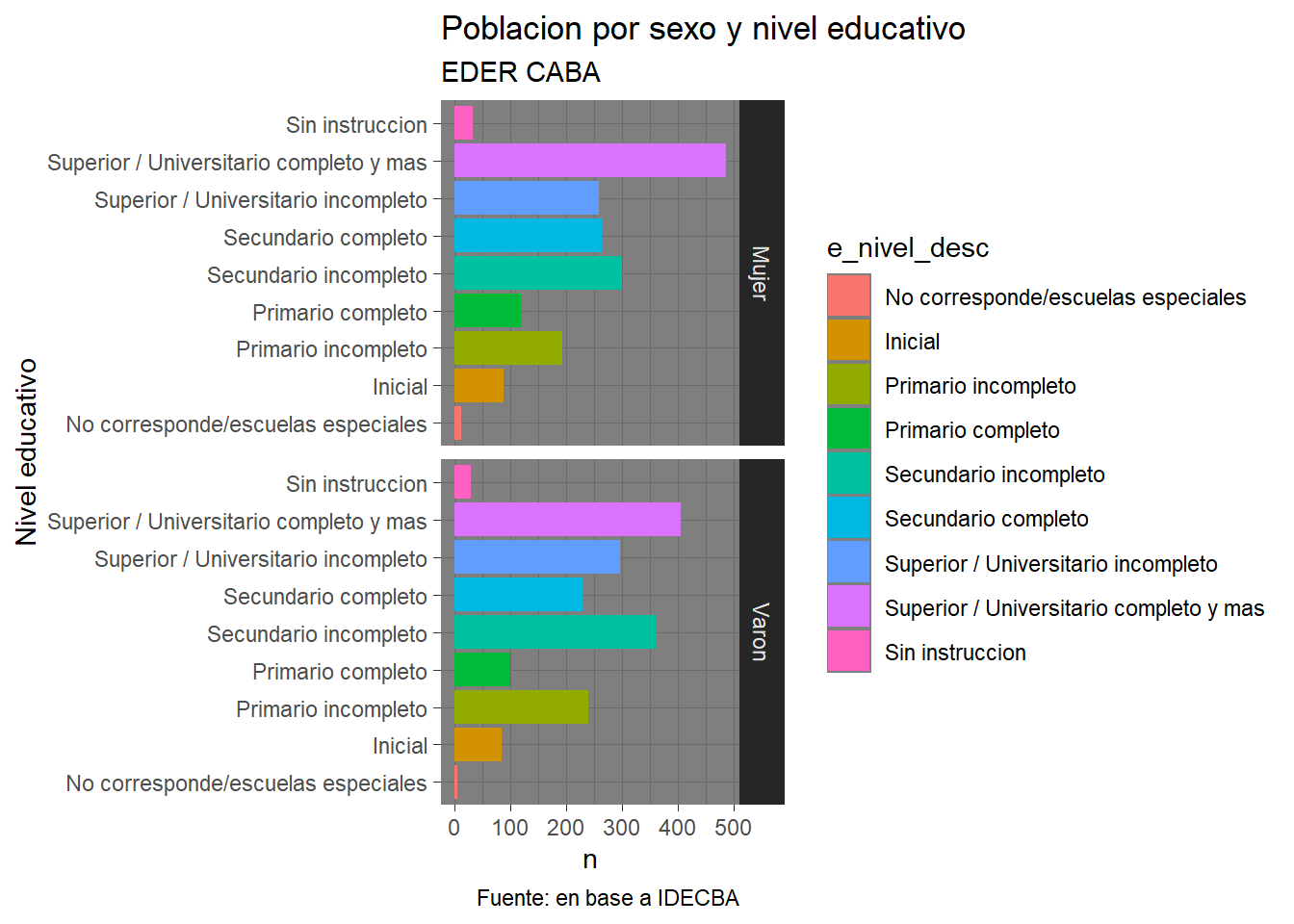
¿Podríamos mostrar las distribuciones al interior del total? ¡Claro!
g_ned_sexo_stack <- eder_ned_sexo_long %>%
ggplot(aes(x = Sexo, y = n, fill = e_nivel_desc))+
geom_col(stat = "identity")+ # con este parámetro, se apilan
scale_y_continuous(labels = abs) +
labs(y = "n",
x = "Nivel educativo",
title = "Poblacion por sexo y nivel educativo",
subtitle = "EDER CABA",
caption = "Fuente: en base a IDECBA") +
theme_bw()
g_ned_sexo_stack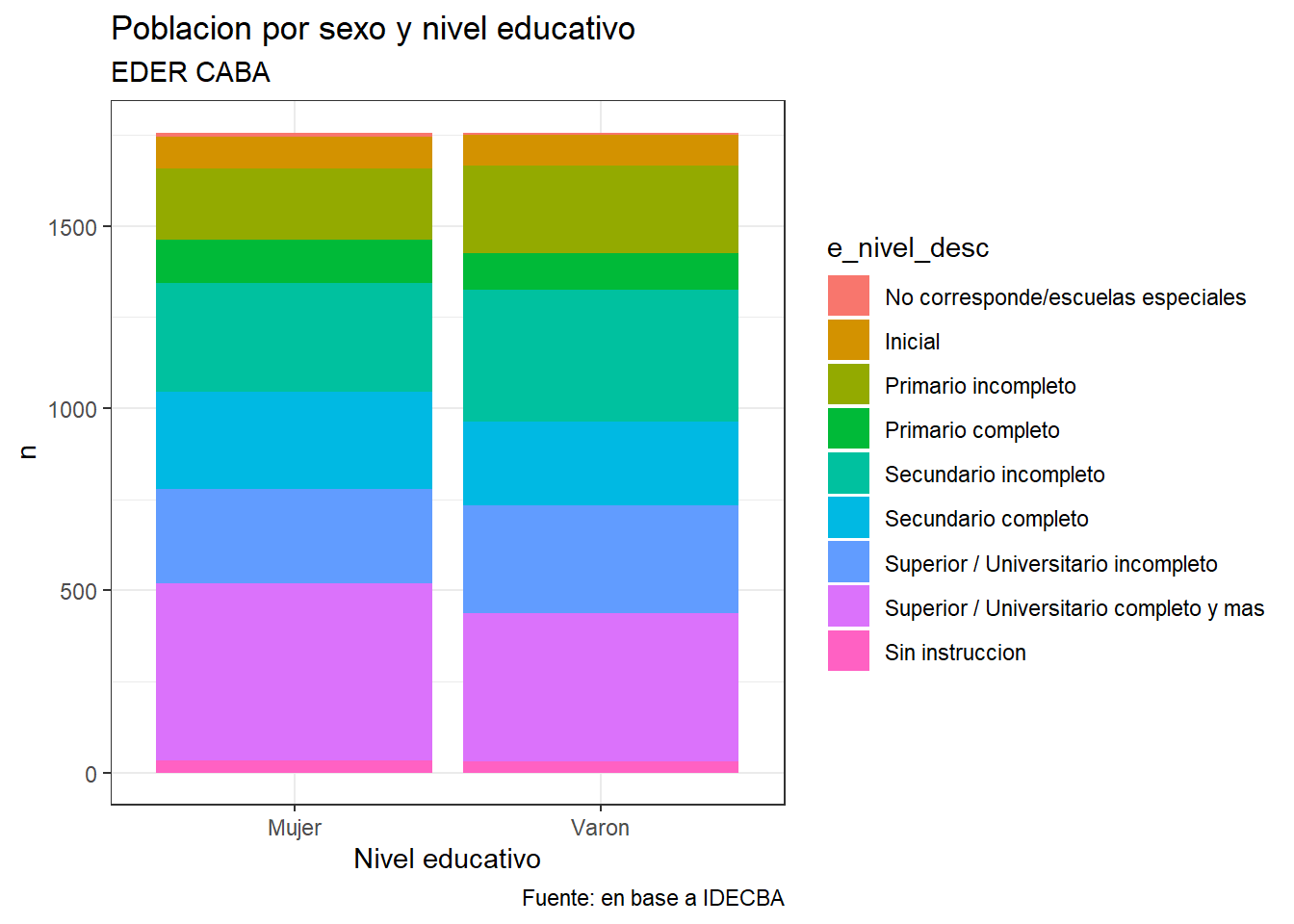
Finalmente, cuando necesites guardar el gráfico, por ejemplo como pdf:
Quizás si fuera un gráfico interactivo… Un bonus track utilzando plotly:
4.3 Lexis
Gráfico útil para representar la dinámica poblacional de ingreso y permanencia en un estadío demográfico según edad, tiempo calendario (o período) y año de nacimiento (o cohorte). Su aplicación más común es en mortalidad, pero es generalizable a la relación exposición~evento con múltiples usos.
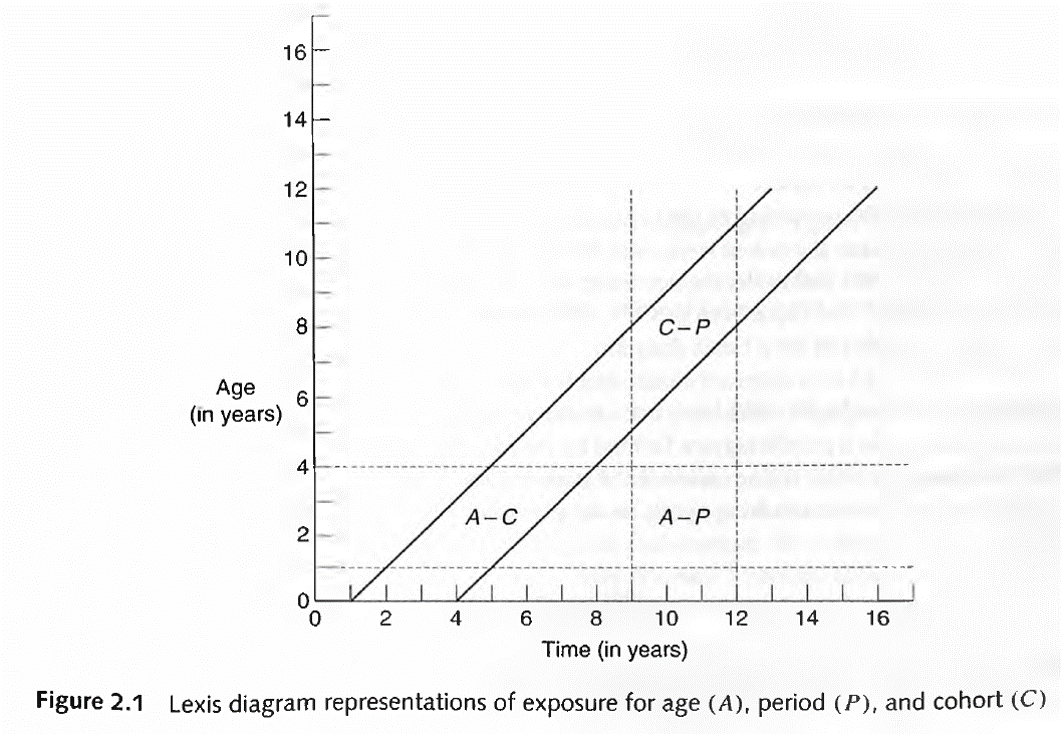
Los elementos principales de análisis son:
- Líneas de vida (45°)
- Segmentos horizontales y verticales
- Superficies
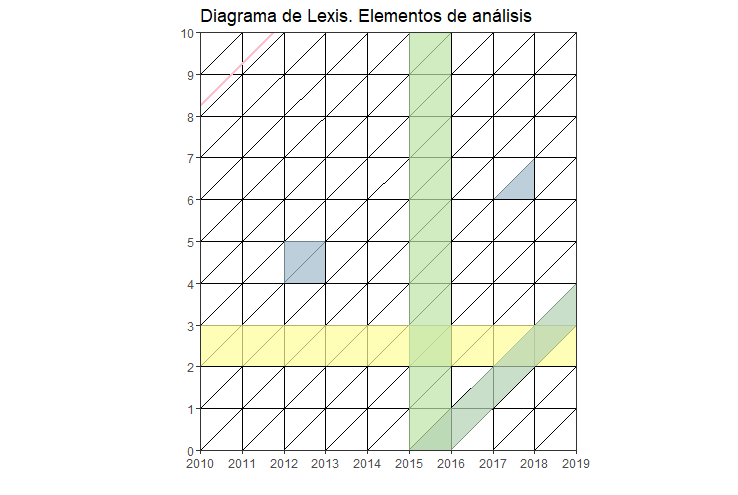
Ejemplos de uso del diagrama utilizando R:
Mortalidad por conflictos armados (replicable):
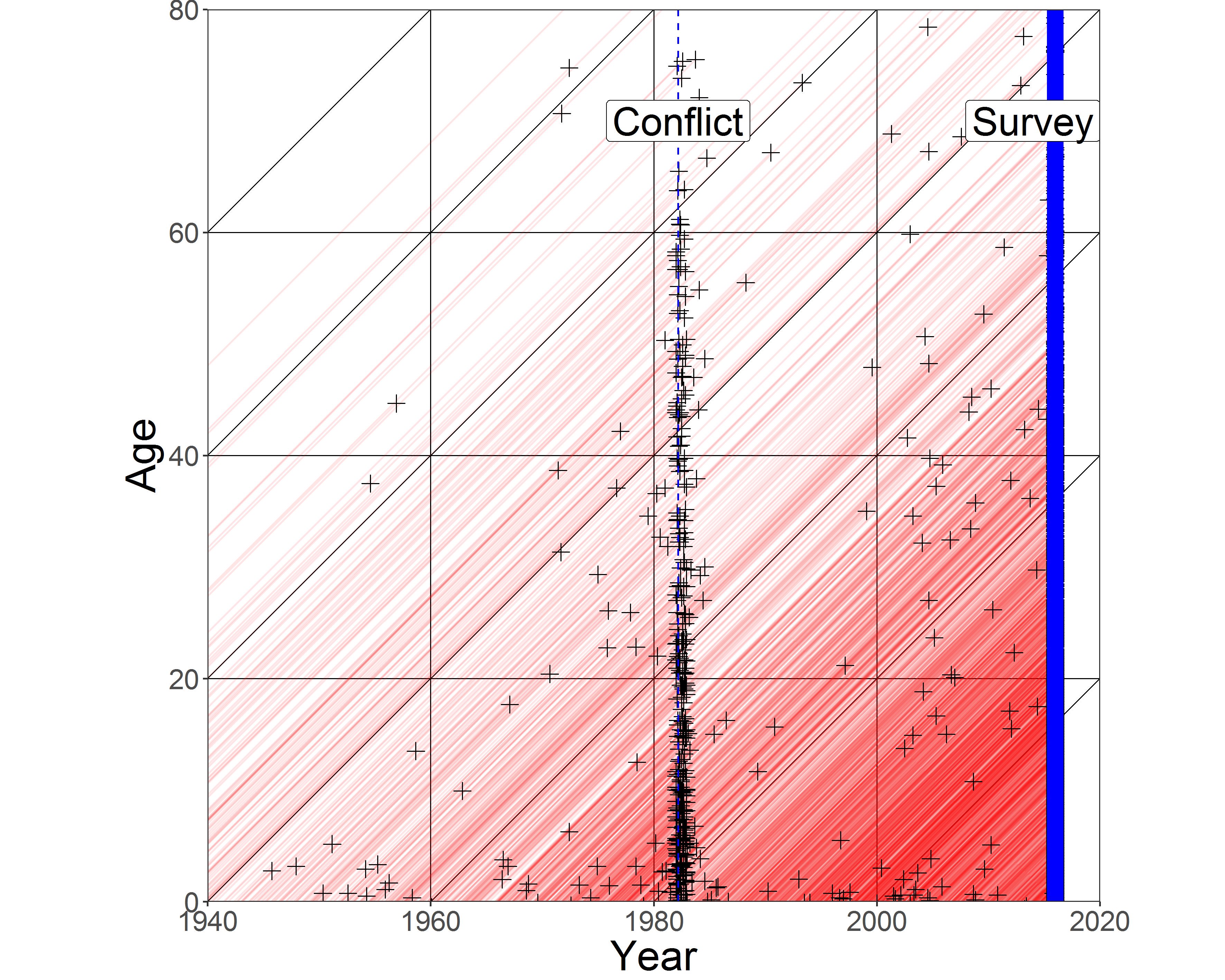 Fuente: Alburez et. al (2019)
Fuente: Alburez et. al (2019)Sobremortalidad masculina en Inglaterra y Gales (1841-2013):
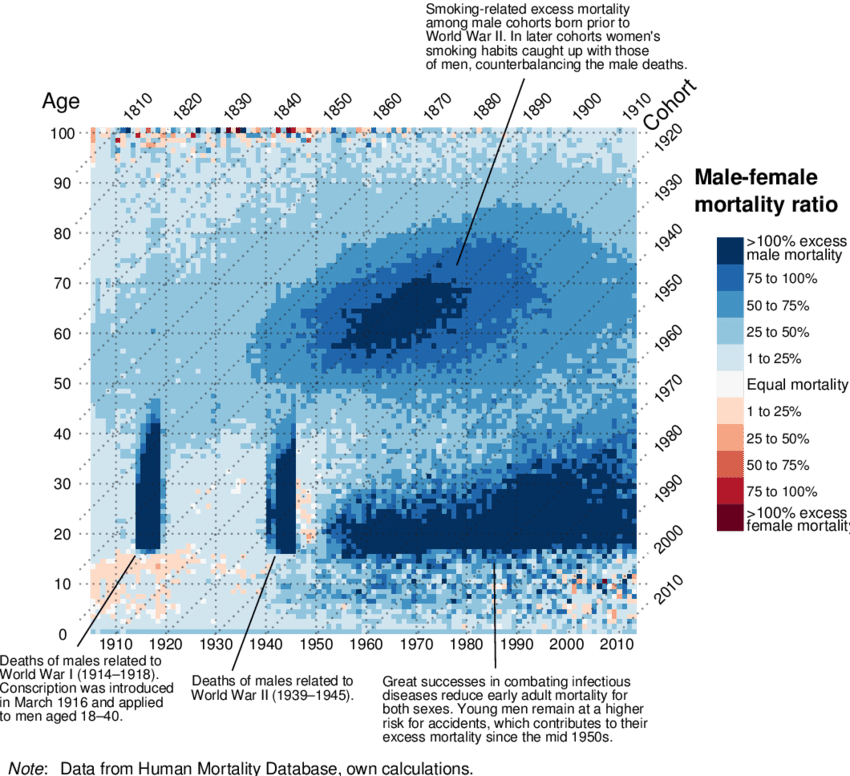 Fuente: Schöley & Willekens (2017)
Fuente: Schöley & Willekens (2017)
Construyamos el diagrama básico del inicio. Para eso haremos uso del paquete LexisPlotR creado por Philipp Ottolinger (gracias Otto). Construyó un set de funciones basadas en ggplot que facilita la creación de diagramas haciéndolo un ejercicio intuitivo.
# install.packages("devtools")
# devtools::install_github("ottlngr/LexisPlotR") #¿Cómo no esta en CRAN?
library(LexisPlotR)
library(tidyverse)
# creamos el objeto "mylexis", definiendo el rango de sus ejes: los primeros 10 años de vida durante 2010-2019. Aquí podemos especificar un "delta" si es que nos interesan los grupos quinquenales.
mylexis <- lexis_grid(year_start = 2010, year_end = 2019, age_start = 0, age_end = 10)
# pintamos la edad 2
mylexis <- lexis_age(lg = mylexis, age = 2)
# pintamos el año 2015, de gris en este caso.
mylexis <- lexis_year(lg = mylexis, year = 2015, fill = "grey")
# Ahora la cohorte 2015
mylexis <- lexis_cohort(lg = mylexis, cohort = 2015)
# Una línea de vida, ¡elemento fundamental del diagrama!
mylexis <- lexis_lifeline(lg = mylexis, birth = "2001-09-23", lwd = 1, colour = "pink")
# Al ser un objeto ggplot, podemos incorporarle atributos, como el título
mylexis <- mylexis + labs(x="Año", y = "Edad",title = "Diagrama de Lexis. Elementos de análisis")
# Para crear polígonos, el autor se basa en la geometría "geom_polygon". Creamos un cuadrado. El orden de los datos es importante. Además podemos crear muchos polígonos al mismo tiempo señalando los grupos.
square <- data.frame(group = c(1, 1, 1, 1),
x = c("2012-01-01", "2012-01-01", "2013-01-01", "2013-01-01"),
y = c(4, 5, 5, 4))
mylexis <- lexis_polygon(lg = mylexis, x = square$x, y = square$y, group = square$group)
# Creamos el triángulo. ¿A qué refiere?
triangle <- data.frame(group = c(1, 1, 1),
x = c("2017-01-01", "2018-01-01", "2018-01-01"),
y = c(6, 6, 7))
mylexis <- lexis_polygon(lg = mylexis, x = triangle$x, y = triangle$y, group = triangle$group)
# Imprimimos!
mylexis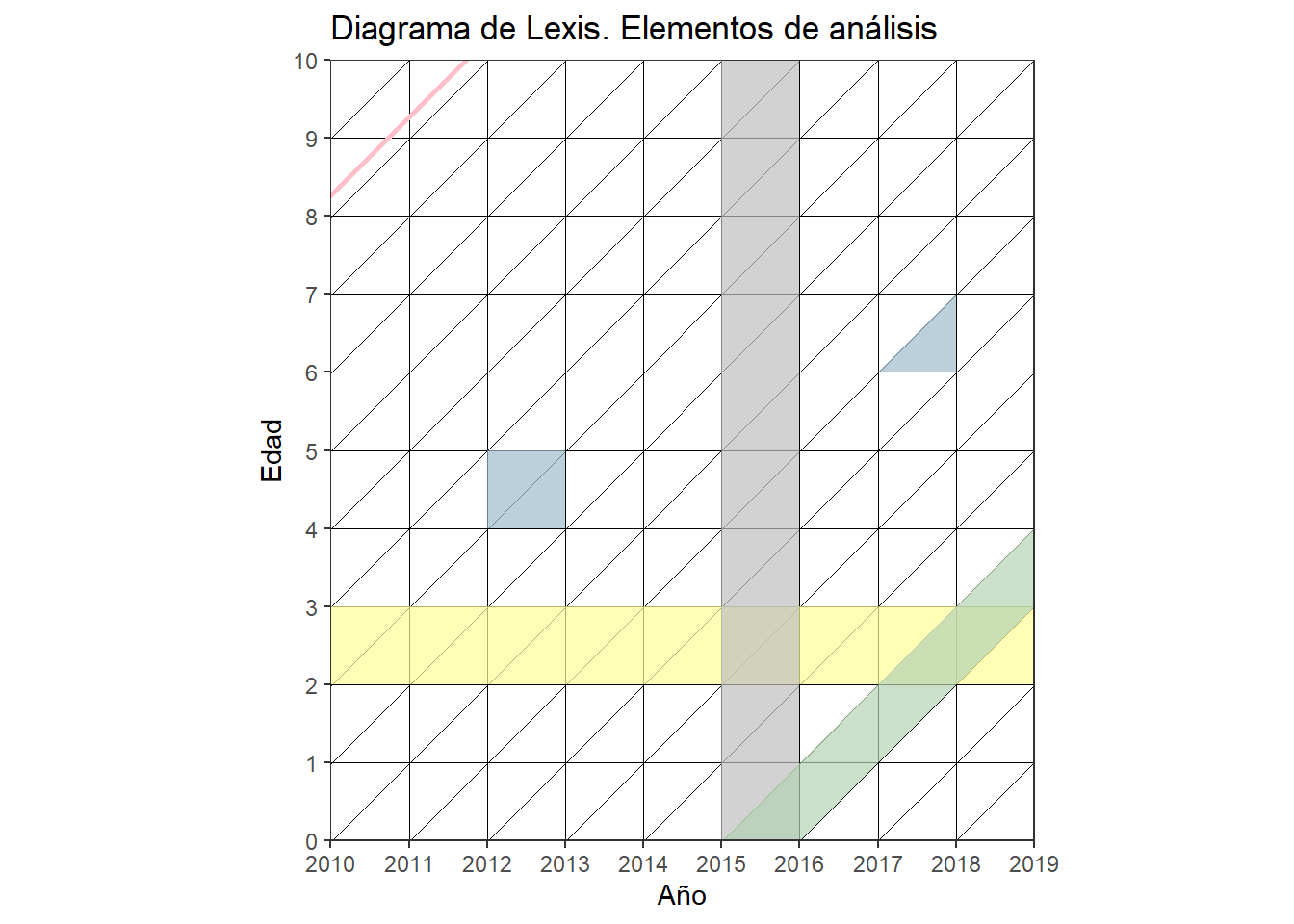
Adicionalmente, si trabajamos con población donde la exposición puede ser truncada (por derecha o izquierda), podemos graficar líneas de vida que respeten ese comportamiento. Tambien podemos señalar con una cruz la ocurrencia del evento de salida que se estudia.
# ingresa al estudio sobre deserción escolar en el año 2012 un niño/a nacido en el año 2005, abandonando el colegio a los 9 años de edad:
mylexis <- lexis_lifeline(lg = mylexis,
birth = "2005-09-23",
entry = "2012-06-11", exit = "2015-02-27", lineends = T,
colour = "red")
mylexis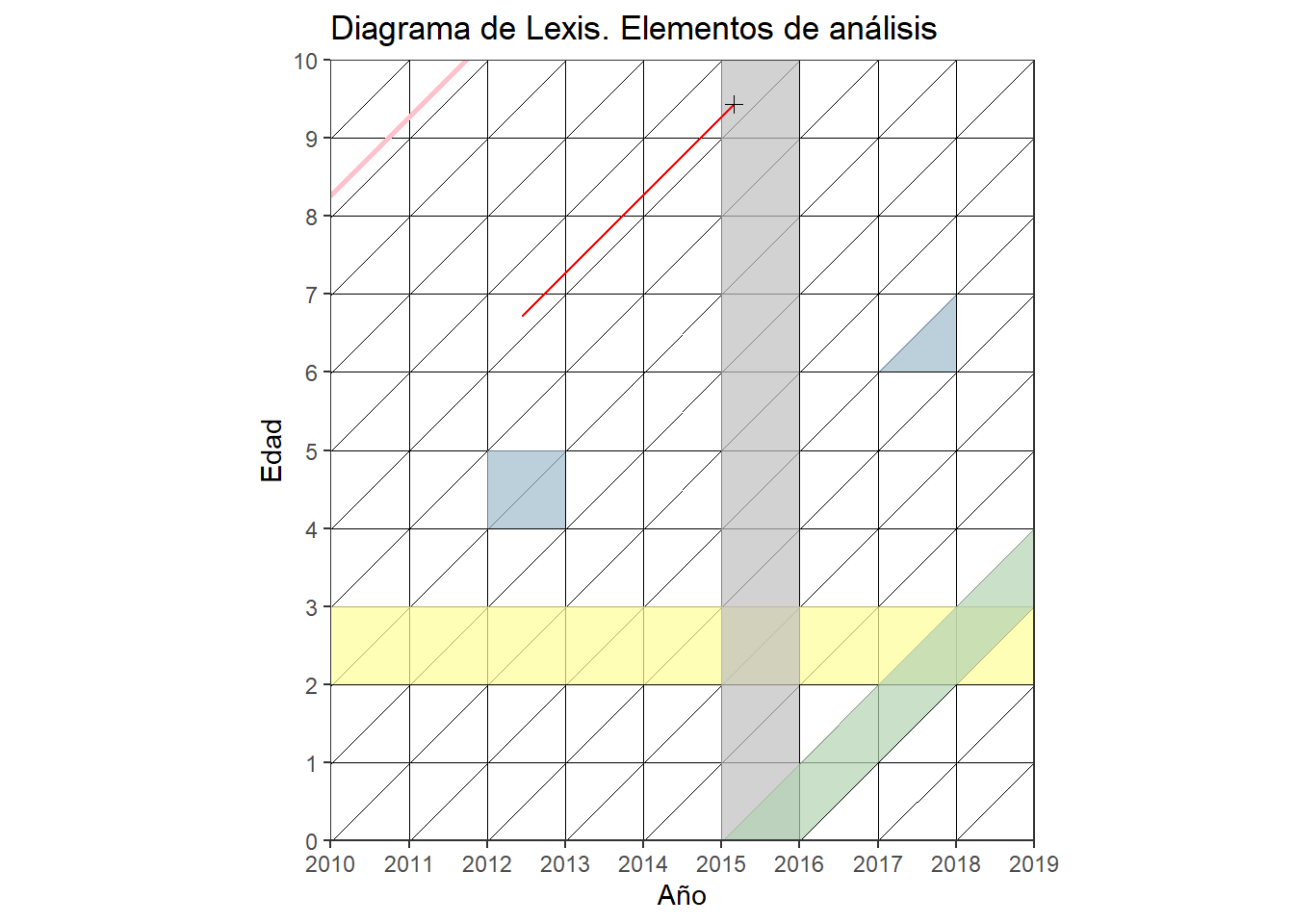
Pero atrás de esto esta ggplot!
ggplot() +
geom_segment(aes(x = 2011, y = 5, xend = 2015, yend = 9), color = "red", size=1)+ # debería ir al final
geom_point(aes(x=2015, y=9),shape=4,size=2,col=4)+
coord_equal() +
scale_x_continuous(breaks=2010:2020, expand = c(0,0)) +
scale_y_continuous(breaks=0:10, expand = c(0,0)) +
geom_vline(xintercept = 2010:2020, color="grey", size=.15, alpha = 0.8) +
geom_hline(yintercept = 0:10, color="grey", size=.15, alpha = 0.8) +
geom_abline(intercept = seq(-2020, -2000, by=1), slope = 1, color="grey", size=.15, alpha = 0.8)+
labs(x="Año", y="Edad",title = "Dieagrama de Lexis c/ ggplot")+
theme_minimal()+
theme(panel.grid.major = element_line(colour = NA),
panel.grid.minor = element_line(colour = NA),
plot.background = element_rect(fill = "white",
colour = "transparent"))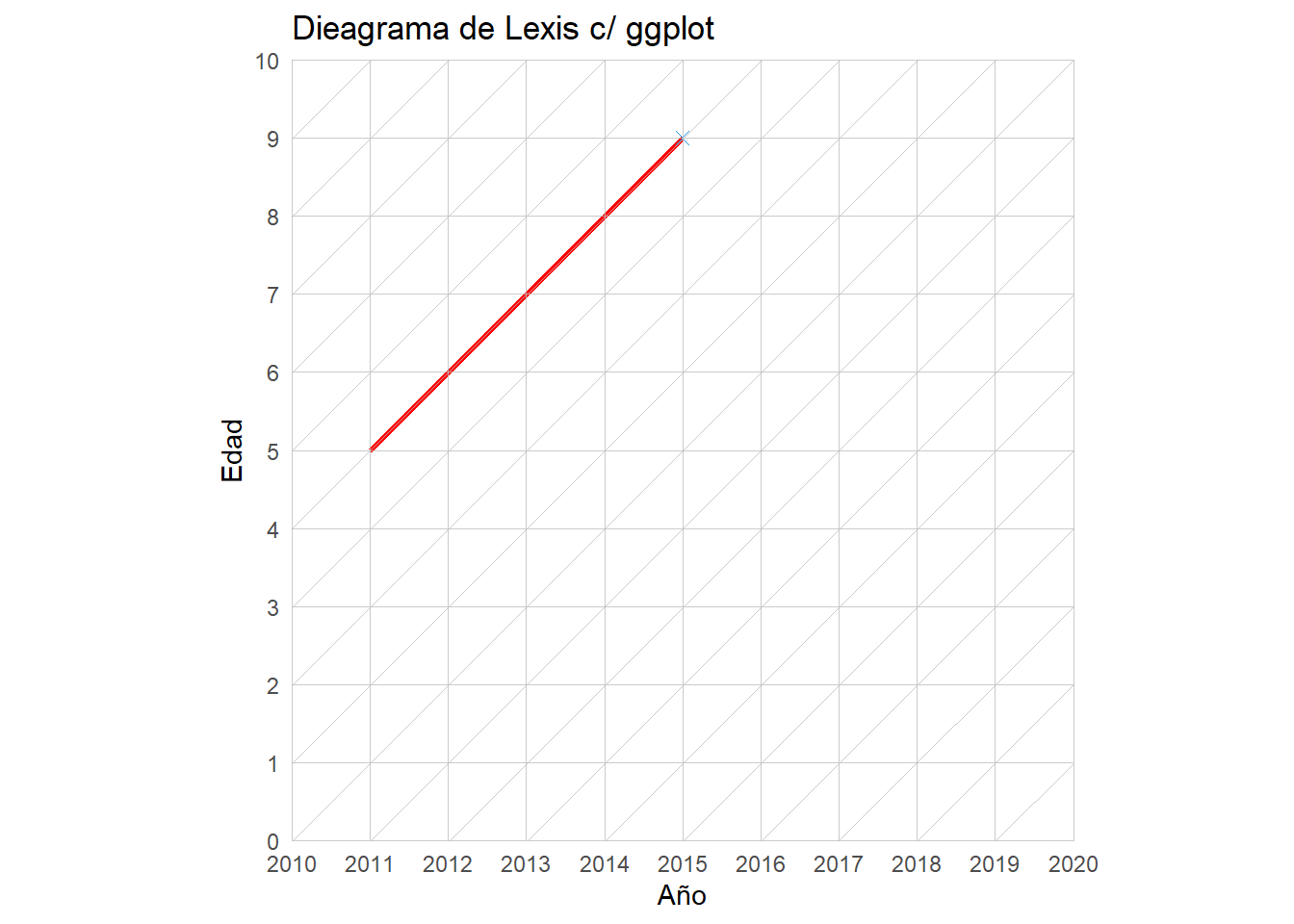
Para incluir triángulos y cuadriláteros se utiliza geom_polygon. Para dar superficie, de manera similar al gráfico Scholey & Willekens (2017), se utiliza geom_tile. Para esto último un gran tutorial de Tim Riffe.
4.4 Estudio de cohorte: un ejemplo
Construyamos una tabla de cohorte a partir de un grupo de estudiantes que iniciaron con 3 años de edad (cumplida) su trayectoria educativa el 3/3/2000, y donde la salida es dejar el estudio (sea cual sea el nivel alcanzado) por primera vez antes de retomar o abandonar definitivamente (¿en este contexto qué sería \(\omega\)?). Simulemos el comportamiento de 1000 chicas/os con distribución uniforme de la edad y distribución gamma del tiempo de permanencia:
set.seed(100) # qué es esto?
clase <- data.frame(edad_inicio = runif(n = 1000, min = 3, max = 4),
tiempo_exp = rgamma(n = 1000, shape = 40, rate = 2)) %>%
mutate(edad_salida = edad_inicio + tiempo_exp)¿Cómo luce?
ggplot(clase, aes(x=edad_salida)) +
geom_histogram(fill=2, alpha=.5) +
labs(x = "Edad de salida", y = "Densidad",
title = "1000 casos simulados de edad de primer salida educativa")## `stat_bin()` using `bins = 30`. Pick better value `binwidth`.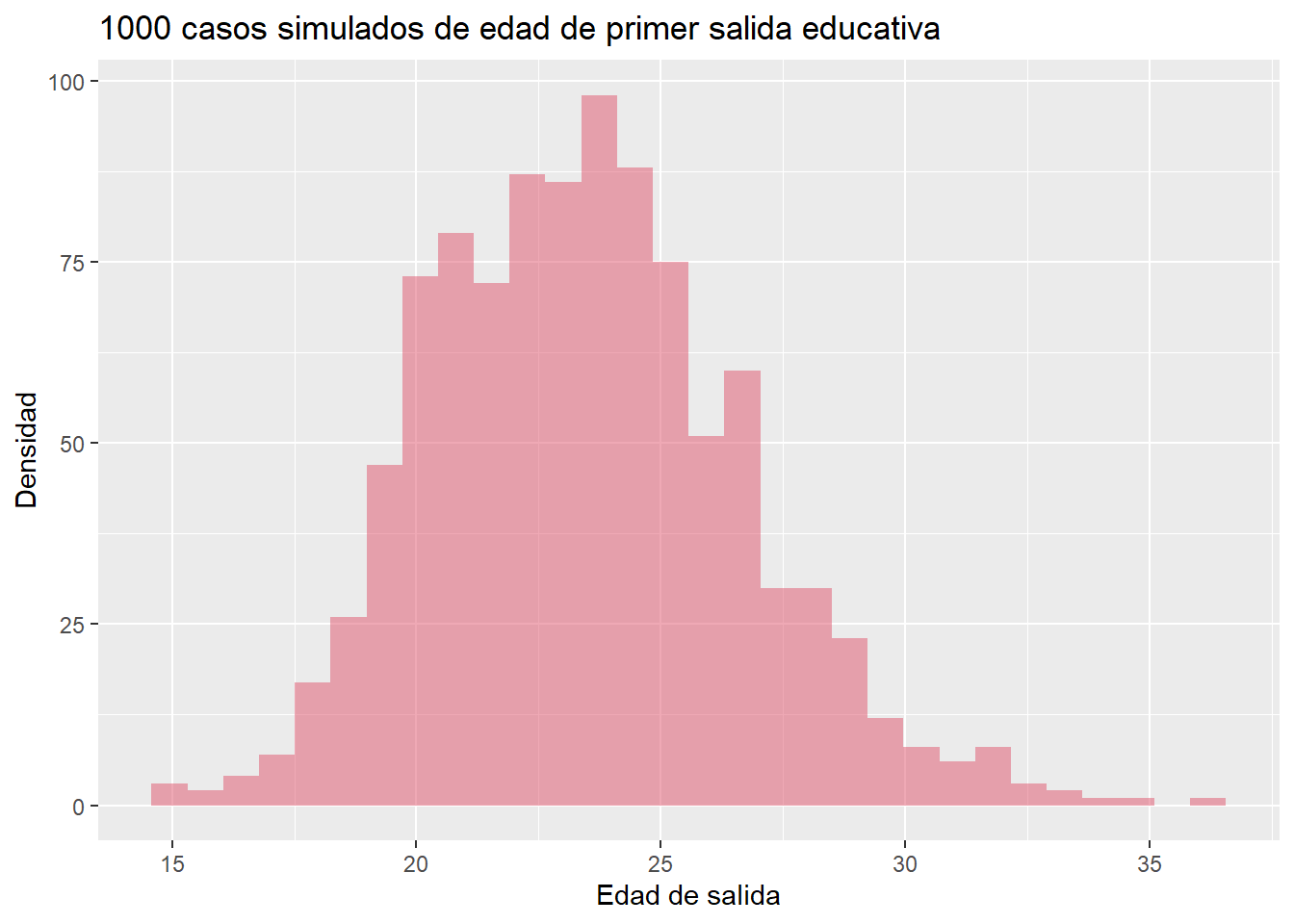
Construyamos el diagrama de Lexis de esta cohorte utilizando LexisPlotR:
# una forma de moverse en el tiempo: a una fecha restarle cantidad de días
clase$nacimiento <- as.Date("2000-03-03") - clase$edad_inicio * 365.25
clase$salida <- clase$nacimiento + clase$tiempo_exp * 365.25
# creo Lexis
mylexis <- lexis_grid(year_start = 2000, year_end = 2025, age_start = 3, age_end = 30)
mylexis <- lexis_lifeline(lg = mylexis,
birth = clase$nacimiento,
entry = "2000-03-03",
exit = clase$salida, lineends = T,
colour = 4, alpha = 1/3) +
theme(axis.text.x = element_text(angle = 90, vjust = 0.5, hjust=1))
mylexis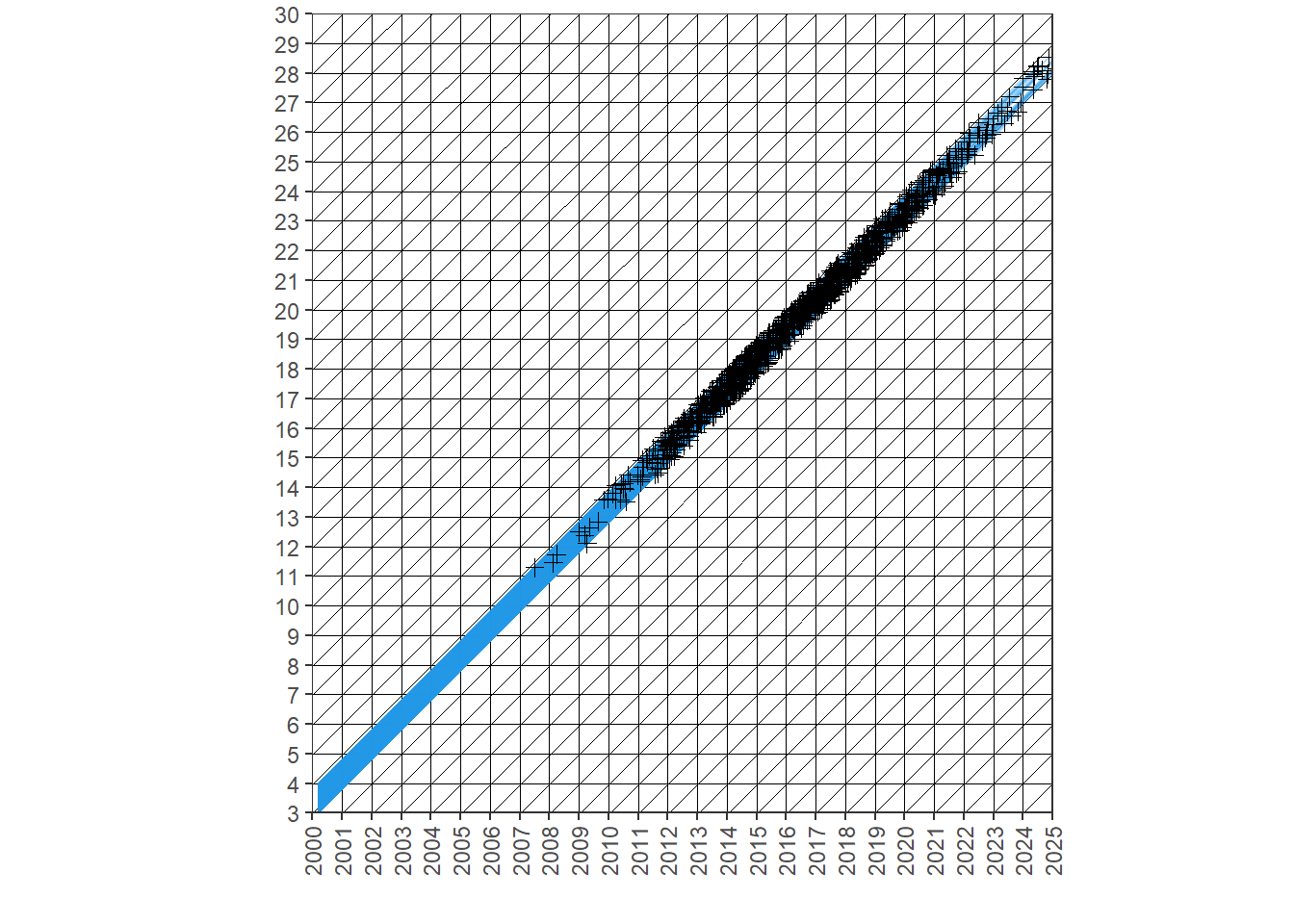
¿Por qué las líneas no empiezan justo en 2000? Estimemos algunos parámetros básicos:
# la edad media de salida
mean(clase$edad_salida)
# la probablidad de "sobrevivir" a la edad 18
clase %>% filter(edad_salida >= 18) %>% summarise(n()/nrow(clase))
# probablidad de abandonar en los 20 años cumplidos
clase %>% filter(edad_salida >= 20 & edad_salida < 21) %>% summarise(n()/nrow(clase))## [1] 23.4662
## n()/nrow(clase)
## 1 0.974
## n()/nrow(clase)
## 1 0.1034.4.1 Actividad
Crear un diagrama de Lexis e incluir en rojo las líneas de vida de las 3 personas más jóvenes que conozcas.
Considerando el ejercicio de permanencia escolar:
Calcula la probabilidad de “salir” luego de los 30 años?
Cambia la semilla del ejercicio previo (
set.seed()) por cualquier entero que desees y repite el ejercicio. ¿Hay diferencias? ¿Por qué?
4.5 Reproduciendo
El covid19 puso en primera plana el análisis demográfico, especialmente en el impacto que tuvo en \(e_0\) ¿No te parece genial la figura 1 del paper Quantifying impacts of the COVID-19 pandemic through life-expectancy losses: a population-level study of 29 countries de Aburto y Otros (2021)? Podemos recrearlo gracias a que los autores lo permiten aquí, haciendo su paper reproducible. De paso vamos viendo funciones nuevas. Iremos comentando los pasos:
# necesitamos algunas librerías que no tenemos aún. Si no las tenés debes instalarlas primero
#install.packages("hrbrthemes")
library(hrbrthemes)
# cargar la data
df_ex_ci <- readRDS("Data/df_ex_ci.rds")
head(df_ex_ci)## # A tibble: 6 × 19
## code sex year age ex ex_diff ex_diff_1519 ex_2015 ex_2019 ex_2020
## <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 AT Female 2020 0 83.6 -0.671 0.490 83.8 84.3 83.6
## 2 AT Female 2020 1 82.8 -0.650 0.447 83.0 83.4 82.8
## 3 AT Female 2020 2 81.8 -0.648 0.439 82.0 82.5 81.8
## 4 AT Female 2020 3 80.9 -0.652 0.442 81.1 81.5 80.9
## 5 AT Female 2020 4 79.9 -0.657 0.449 80.1 80.5 79.9
## 6 AT Female 2020 5 78.9 -0.662 0.455 79.1 79.6 78.9
## # ℹ 9 more variables: name <fct>, code_hmd <chr>, rank_e0f19 <int>,
## # rank_d0m20 <int>, ex_diff_1920_q025 <dbl>, ex_diff_1920_q975 <dbl>,
## # ex_avgdiff_pre2020 <dbl>, ex_avgdiff_pre2020_q025 <dbl>,
## # ex_avgdiff_pre2020_q975 <dbl># crear el gráfico
fig_1 <- df_ex_ci %>%
# primero factorizar según el número actual de la variable
mutate(name = name %>% fct_reorder(rank_e0f19)) %>%
# me interesa ciertas edades
filter(age %in% c(0, 60)) %>%
# si hay alguna NA sacar la fila
drop_na(name) %>%
# una forma de seleccionar
transmute(name, sex, age,ex_2015, ex_2019, ex_2020 = ex) %>%
# hete aquí un pivot...
pivot_longer(cols = ex_2015:ex_2020,
names_to = "year", values_to = "ex", names_prefix = "ex_") %>%
mutate(age = age %>% as_factor()) %>%
# ggplot
ggplot()+
# un color por sexo, una forma por año
geom_point(aes(x = ex, y = name, color = sex, shape = year))+
# separar un poco con líneas grises
geom_hline(yintercept = seq(2, 28, 2), size = 5, color = "#eaeaea")+
# personalizar qué formas, tamaños y colores usar
scale_shape_manual(values = c(124, 43, 16))+
scale_size_manual(values = c(4, 4, 1.5))+
scale_color_manual(values = c("#B5223BFF", "#64B6EEFF"))+
# donde colocar los nombres de los ejes
scale_y_discrete(position = "right")+
scale_x_continuous(position = "top")+
# facet por edad, sin respetar escala
facet_grid(~age, scales = "free_x")+
# sin leyenda, líneas horizonatales y demases
theme(
legend.position = "none",
panel.grid.major.y = element_blank(),
panel.grid.minor.y = element_blank(),
strip.text = element_blank(),
panel.spacing.x = unit(2, "lines"),
axis.text.y = element_text(face = 2))+
# etiquetas de ejes
labs(x = "Life expectancy, years",y = NULL)
# guardar
ggsave(fig_1, filename = "fig-1.pdf",
width = 6, height = 4.5, device = cairo_pdf)Las etiquetas en referencia a la edad y la formas de los puntos las realizaron posteriormente con annotate, una función que permite incluir (encima) texto u otras figuras geométricas en el plano.
4.6 Otras visualizaciones
Samuel Preston (1975) encontró una relación matemática (al menos) a nivel de país entre la esperanza de vida al nacer y el producto per cápita. Veamos que ocurre específicamente en el Continente Americano utilizando el paquete de datos gapminder.
library(gapminder)
p <- ggplot(filter(gapminder, continent == "Americas"),
aes(x = gdpPercap, y = lifeExp))
p + geom_point() # dispersión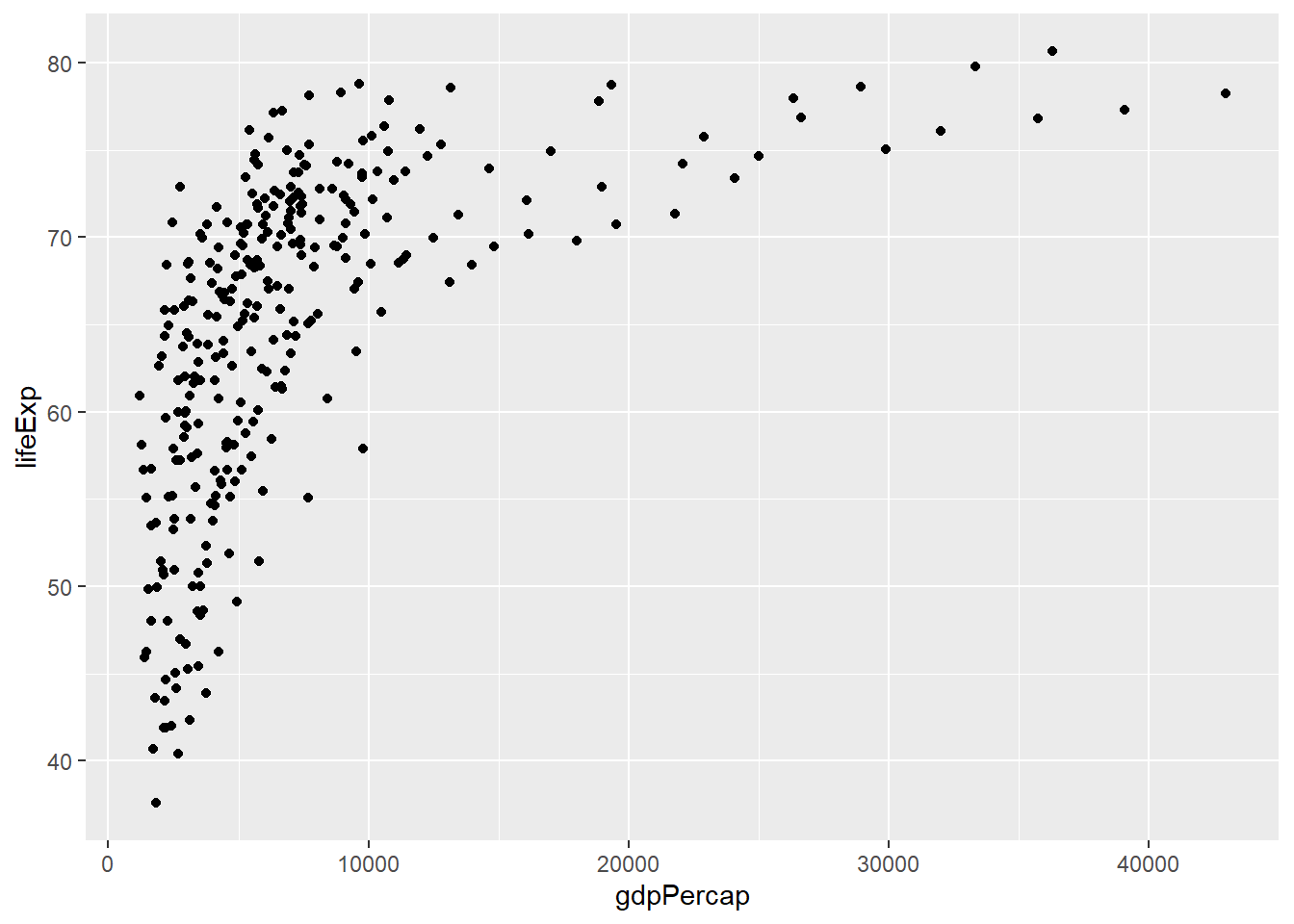
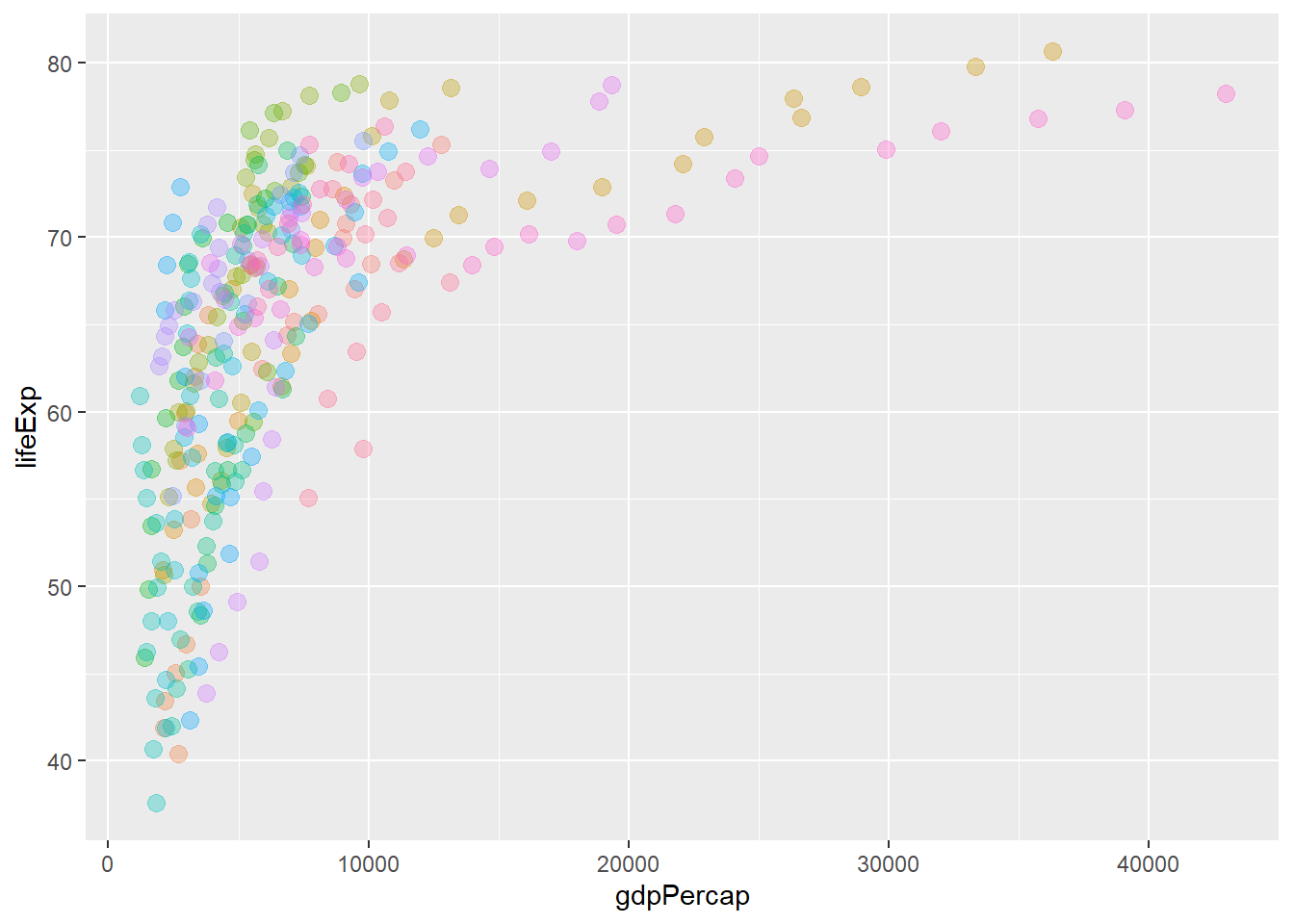
p1 <- p + geom_point(aes(color = country, size = pop), alpha = 1/3) # fijate el lugar de size
p1 <- p1 + geom_smooth(lwd = 1, lty = 2, color = 2, se = FALSE)
p1## `geom_smooth()` using method = 'loess' and formula = 'y ~ x'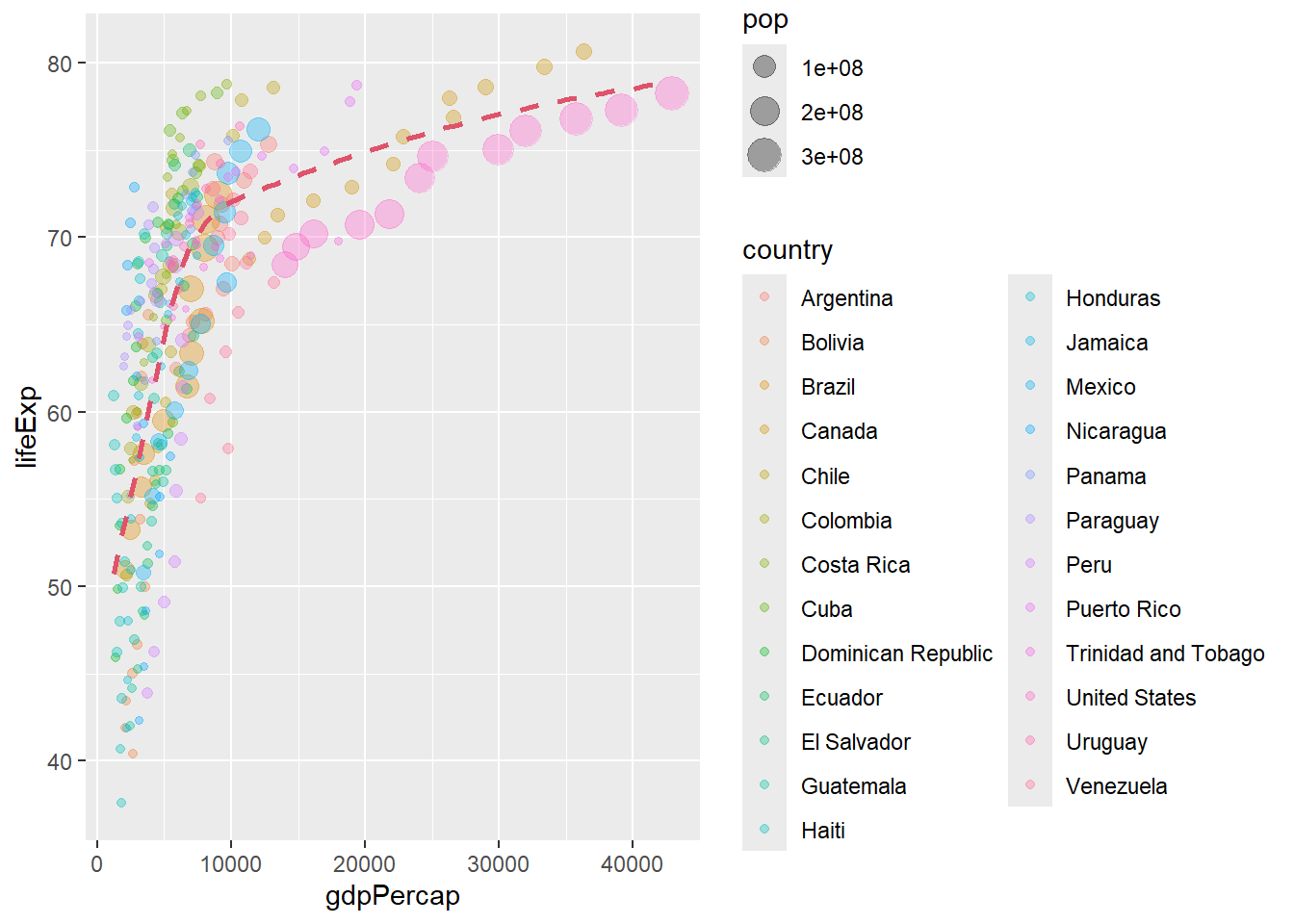
p + geom_point(alpha = 1/3, size = 3) + facet_wrap(~ country) +
geom_smooth(lwd = 1.5, se = FALSE) +
theme_void()## `geom_smooth()` using method = 'loess' and formula = 'y ~ x'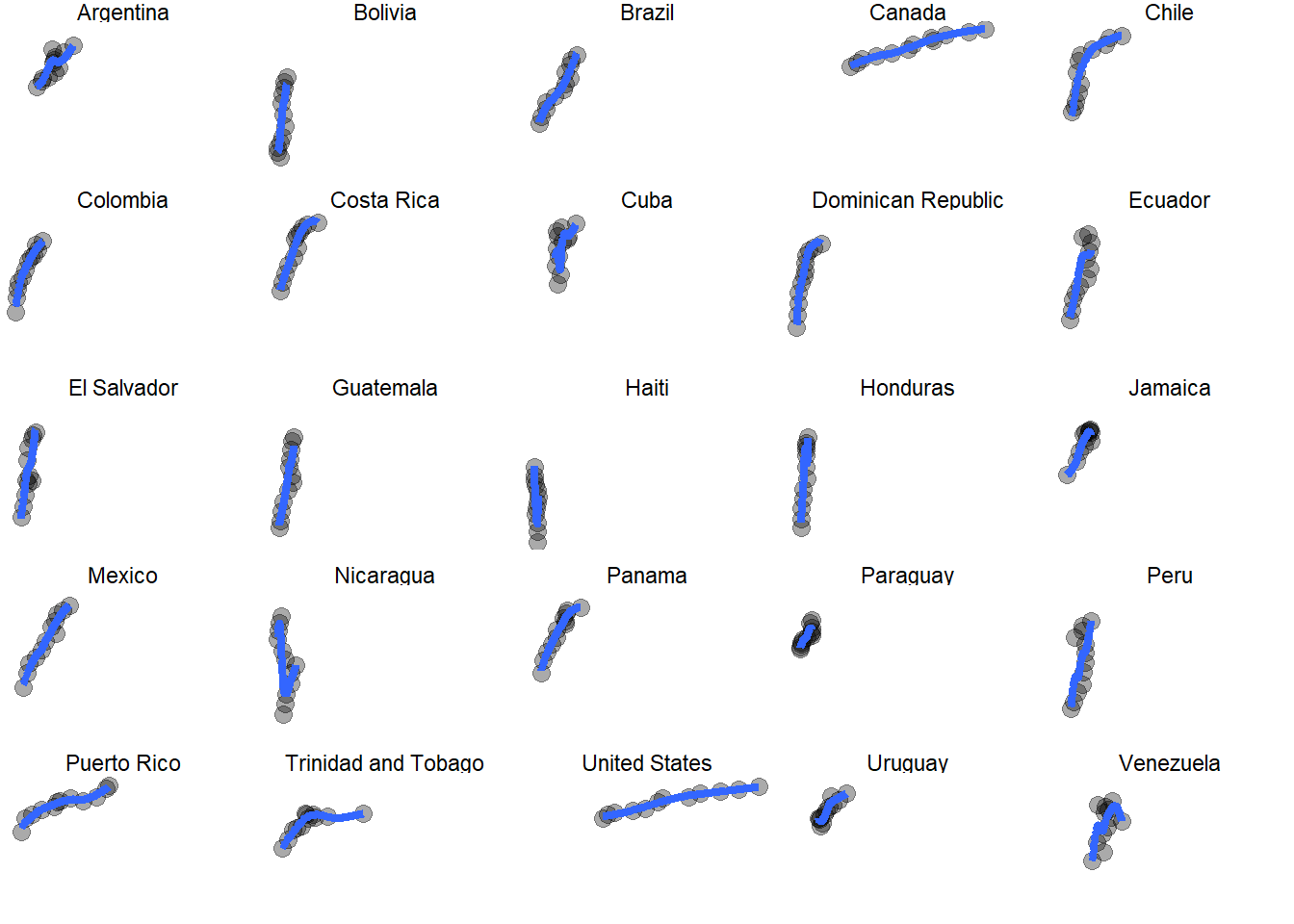
¿Componer gráficos en una sola hoja? (tomado de Jenny Bryan)
##
## Adjuntando el paquete: 'gridExtra'## The following object is masked from 'package:dplyr':
##
## combinep2 <- ggplot(filter(gapminder, year==1952 & continent!="Oceania"), aes(x = lifeExp, color = continent)) + geom_density()
p3 <- ggplot(filter(gapminder, year==2007 & continent!="Oceania"), aes(x = lifeExp, color = continent)) + geom_density()
grid.arrange(p2, p3, nrow = 2, heights = c(0.5, 0.5))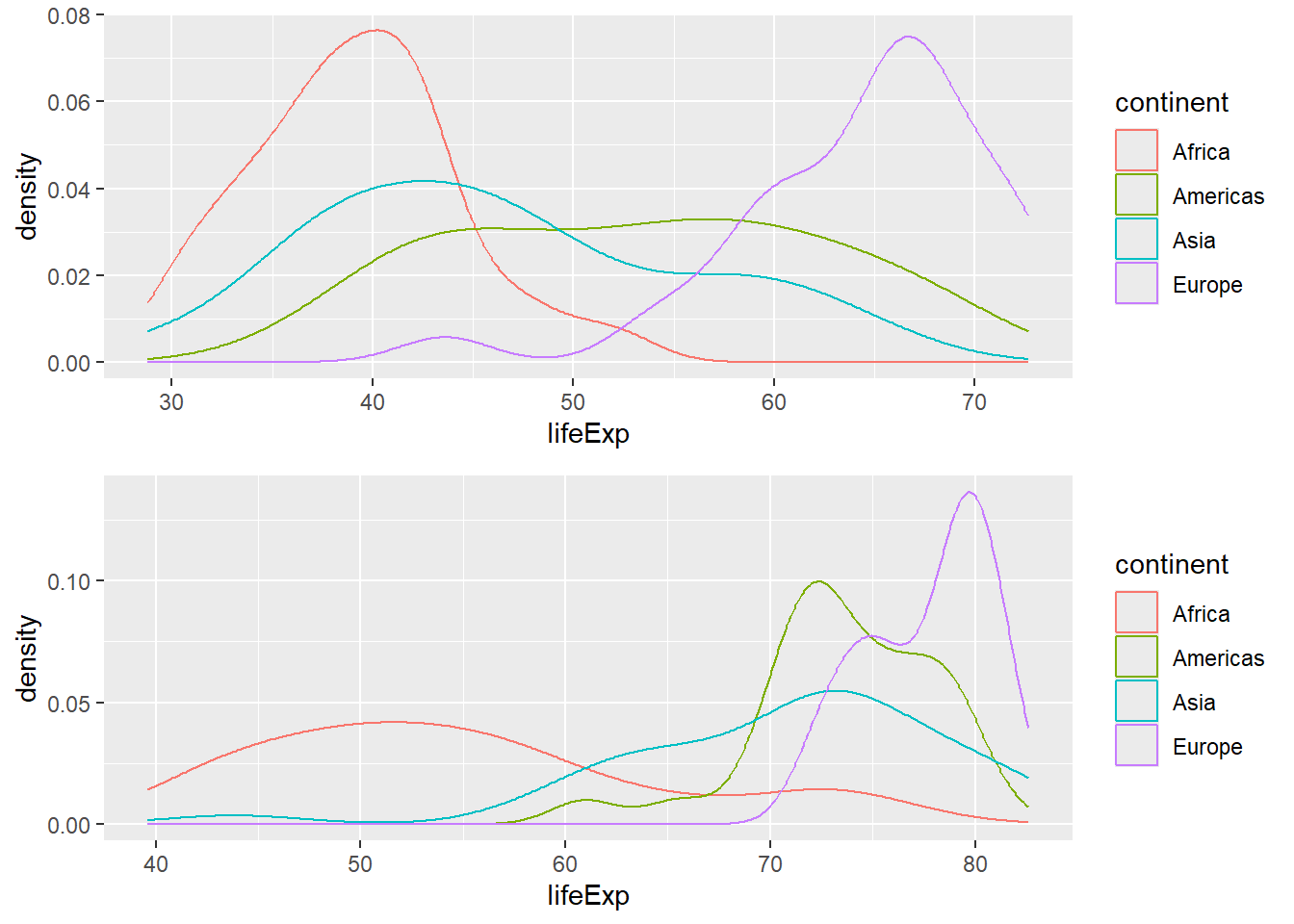
¿Qué problemas encuentras en este arreglo?
4.7 Material adicional (y no tanto)
Sí, hay un libro.
Estas notas fueron hechas siguiendo principalmente The Hitchhiker’s Guide to Ggplot2 y el curso de Jenny Bryan.
Hulíková Tesárková & Kurtinová. Application of “Lexis” Diagram: Contemporary Approach to Demographic Visualization and Selected Examples of Software Applications.
Rau, R., Bohk-Ewald, C., Muszyńska, M. M., & Vaupel, J. W. (2018). Visualizing Mortality Dynamics in the Lexis Diagram. Springer. doi: 10.1007/978-3-319-64820-0
