3 dplyr
3.1 Lectura de datos
En la unidad previa caracterizamos un hogar. En esta unidad trabajaremos la base de datos de la Encuesta Demográfica Restropectiva (EDER) que se realizó en CABA en el año 2019. Esta incluye cuatro bases:
Personas: cada registro es una persona e incluye y caracteriza a todas las personas que componen los hogares entrevistados.
Retro: cada registro representa un año calendario en que suceden los distintos eventos de la persona entrevistada seleccionada para contestar la encuesta retrospectiva.
Retro cónyuge: cada registro representa un año calendario de la persona entrevistada en relación a cada uno de los cónyuges declarados por la persona entrevistada.
Retro hijos: cada registro representa un año calendario de la persona entrevistada en relación a cada uno de los hijos declarados por el entrevistado.
Vamos a empezar trabajando con la base personas. Para esto recorreremos distintos paquetes de la familia tidyverse, cuyo fin es generar un entorno de herramientas especializada en el análisis de datos.
Pero primero haremos un repaso por las distintas opciones de lectura de datos que nos ofrece R. Creemos una carpeta que se llame “data”, donde dejemos las bases de datos enviadas. Verifiquemos que tengamos todas las bases de datos correspondientes en la carpeta “data” mediante list.files().
Para mostrar las diversas formas de lectura de datos que ofrece R nos valdremos de la Encuesta Permanente de Hogares (EPH), un operativo estadístico llevado adelante por INDEC con motivo de caracterizar la evolución de los mercados de trabajo de 31 aglomerados urbanos del país. Por ejemplo, si quisiéramos leer la base de datos del tercer trimestre de 2024, ¿qué opciones tenemos? ¿Qué opciones nos dan? Es muy importante aquí que la ruta del archivo a leer sea a partir de la posición de trabajo actual (recuerda getwd()) y hacia donde apuntar (puedes utilizar el predictor con tab luego de generar comillas).
- Texto plano con separador
;
# R base trae funciones para la lectura de datos, las más comunes son "read.table" y "read.csv"
EPH_3t24_txt <- read.table(file = "data/usu_individual_T324.txt", header = T, sep = ";")
head(EPH_3t24_txt[,1:5])- Una planilla excel con extensión
xlsoxlsx, seleccionando la pestaña (y el rango de celdas si queremos). Aprovechemos a leer la documentación de esta función:?read_xlso desde el tab de paquetes en RStudio.
library(readxl) # pertenece a tidyverse
EPH_3t24_xls <- read_xlsx(path = "data/usu_individual_T324.xlsx", sheet = 1)
head(EPH_3t24_xls)Nos interesa el rango “B3:C5”:
- También podemos leer extensiones .dbf, SAS, SPSS, Stata, etcéteras… gracias a la librería foreign. Por ejemplo, una base de datos del entorno SPSS de la EPH del primer trimestre de 2019:
- En la primer unidad dijimos que un paquete es compartir , y es lo que hace a una comunidad dinámica. Ya existe un paquete llamado eph. Permite no solo leer las bases de datos trimestre a trimestre, sino funciones para obtener muchos resultados y panelear olas. ¿Qué warning nos da?
3.1.1 Acceso a archivos no locales
También podemos leer un archivo online, por ejemplo la base de microdatos de DEIS (Dirección de Estadísticas e Información en Salud) de defunciones para los años 2005-2022. Vayamos a la web de Datos Argentina del Ministerio de Salud de la Nación, específicamente para Estadísticas Vitales, y veamos cómo obtener la dirección en la web con click derecho sobre el enlace. Tiene extensión .csv. Utilicemos la función read.csv comentada anteriormente, pero en vez de apuntar a un archivo local hagámoslo al url:
url <- "http://datos.salud.gob.ar/dataset/27c588e8-43d0-411a-a40c-7ecc563c2c9f/resource/ccf688f4-db38-4b0f-86f3-c99341ac52ad/download/defunciones-ocurridas-y-registradas-en-la-republica-argentina-entre-los-anos-2005-2022.csv"
DEf05_22 <- read.csv(url, header = T)
head(DEf05_22)Otras opciones posibles:
Paquete con datos: por ejemplo
Con conexión a un servidor: podemos acceder a una base de datos SQL Server, Oracle, etc. desde R, leyendo (y editando) sus tablas a partir de las credenciales que tengamos asignadas.
Planillas de google: tenemos la posibilidad de leer y escribir planillas de cálculo en la nube.
API: pedidos online de información a un tercero. Por ejemplo, en su momento twitter (o X ahora), o servicios específicos como el de la OMS.
3.1.2 Actividad
Seguro ya estás cansadx de levantar datos: “Dame la EDER” se escucha murmurar en el aula-zoom.
Trabajaremos con la base de datos personas de la EDER 2019.
Terminemos de afianzar lo visto hasta aquí. Te proponemos resolver/responder:
Chequear que todas las versiones de la base de datos de la eph que leímos tengan la misma cantidad de observaciones (filas).
¿Cuántas muertes fueron registradas en el año 2022? ¿Cuántas de mujeres? ¿Cuántas en la provincia de Santa Fe? Si, necesitas un diccionario de datos, incluido en la web.
Siguiendo con la base de defunciones, estimemos el impacto del exceso de mortalidad del año 2020 y el año 2021 (un número para cada año). Lo definiremos acá como el ratio entre las defunciones de 2020 (o 2021) respecto al promedio de los años 2017-2019.
Leer “eder2019_personas.csv” (por deafult con separador “,”) y guardarlo en un objeto llamado eder_personas_br (br por bruta, aún sin cambios).
Explorar el objeto leído en el punto anterior con lo visto hasta quí, y resaltar aspectos que te llamen la atención.
3.2 dplyr
En la unidad previa caracterizamos un hogar censal. En esta unidad trabajaremos la base de datos de la EDER, con todas las personas encuestadas. Para esto recorreremos algunos paquetes de tidyverse, cuyo fin es generar un entorno integrado de herramientas de análisis de datos.
El paquete dplyr contiene las operaciones más comunes sobre una tabla de datos, y se volvió en los últimos años el paquete más utilizado para este fin.
Una de sus ventajas es el uso del pipe %>% o |>, que concatena operaciones sobre el mismo objeto, verbalizando el proceso, generando fluidez en nuestro razonamiento y mejor entendimiento para un tercero. En vez de f(x) (aplicar una función f() a un objeto x) se tiene x %>% f() (tengo un objeto x y le aplico la función f()):
Previo a comenzar debes haber leído la base de la EDER “eder2019_personas.csv”, haberla inspeccionado brevemente y guardado en un objeto llamado eder_personas_br.
3.2.1 seleccionar (select), renombrar (rename)
Empecemos a trabajar la base de datos de la EDER. Para este ejercicio nos interesa contar con las variables identificación vivienda, hogar, sexo del entrevistado, edad del entrevistado y relación de parentezco con el jefe del hogar. Quedémonos con estas variables mediante la función select:
Podemos realizar la misma operación de la mano de %>%, no necesitando especificar el objeto como argumento:
eder_personas <- eder_personas_br %>% select(id, nhogar, p2, p3b, p4)
# léase: toma el objeto *** y selecciona las columnas ***Si quisiéramos quedarnos solo con las variables de persona podríamos usar starts_with("p") dentro de select. Existen muuuchas más funcionalidades útiles para tablas con muchas columnas.
Nótese que estamos pisando el mismo objeto. Luego podemos renombrar algunas variables para hacer su uso más fluido, mediante rename:
# NuevoNombre = ViejoNombre
eder_personas <- eder_personas %>% rename(id_viv = id, nhog=nhogar, sexo=p2, edad=p3b, rel_par=p4)
# Tambien es posible por posiciónHabrás notado una de sus ventajas: la referencia al nombre de las variables no requiere $ ni “[,]” como en R Base. Podemos condensar lo anterior y hacerlo todo en un paso utilizando %>%. Implica algo super útil: un nuevo %>% supone el objeto ya modificado.
eder_personas <- eder_personas_br%>%
rename(id_viv = id, nhog=nhogar, sexo=p2, edad=p3b, rel_par=p4) %>%
select(id_viv, nhog, sexo, edad, rel_par)¿Lo que estamos generando es un data.frame? ¿Qué es un tibble?
3.2.2 Actividad
Crear un data.frame llamado “eder_personas_edu” que contenga solo las variables que refieran a situación educativa (que tienen un “e” al inicio) de “eder_personas_br”. Podés utilizar la función
starts_with(leer ejemplos).Renombrar las variables de manera que siempre comiencen con “EDU” (en vez de e2 que sea edu2).
Contar la cantidad de columnas mediante
ncol()y corrobar que la cantidad de variables sea menor a las de “eder_personas_br”.
3.2.3 resumir (summarise) por grupos (group_by)
Como analistas nos interesa calcular medidas resumen para poder obtener conclusiones, plantear hipótesis o simplemente conocer más el fenómeno relevado/registrado en los datos. Si quisiéramos saber cuántas personas existen en la base de la EDER podemos realizar un summarise (resumen) de los datos mediante la función n() (contar filas):
BONUS: Si queremos considerar el total de personas expandido (con su ponderador), podemos utilizar la función sum(factor expansión):
Para contar cuántas viviendas fueron relevadas en la base debemos considerar casos con valor distinto en la variable de código de vivienda id_viv:
¿Cuál es el promedio de personas por vivienda dentro de la encuesta?
Cóctel propio
¿De qué forma podríamos hacer lo anterior en R base (nrow,ncol,unique)? La experiencia de usuarix de cada unx define el mix de herramientas que le es más cómodo/útil según la complejidad de cada tarea.
Con summarise podemos aplicar cualquier función resumen sobre los datos. Por ejemplo: ¿Cuál es la edad promedio de las personas? ¿Cual es la edad mínima reportada?
eder_personas_br %>% summarise(edad_media = mean(p3b))
eder_personas_br %>% summarise(edad_min = min(p3b))
eder_personas_br %>% summarise(edad_max = max(p3b))¿Algo te llama la atención?
¿Podemos incluir todo lo anterior en una sola sentencia? ¡Esa es la magia de %>%!
eder_resumen<- eder_personas_br %>%
summarise(n_personas = n(),
n_viviendas = n_distinct(id),
pers_x_viv = n_personas/n_viviendas,
edad_media = mean(p3b),
edad_min = min(p3b)
)Las operaciones principales que podemos realizar mediante summarise las podés ver acá.Pero su utilidad se potencia cuando queremos resumir una variable segmentando por grupos. Esta agrupación antecede a summarise, y requiere ser indicada con group_by incluyendo como argumento las variables para agrupar. Por ejemplo, para conocer la cantidad de personas relevadas por sexo:
Seguro estas cantidades difieren según código de relación de parentesco con lx jefx de hogar. Pero primero podemos agregar la variable que describe los códigos. Carguemos como objeto el diccionario e incorporémoslo:
cod_rel_par <- data.frame(p4 = 1:13,
p4_descripc = c("Jefe/a",
"Cónyuge/pareja",
"Hijo/a",
"Hijastro/a",
"Yerno o nuera",
"Nieto/a",
"Padre/madre/suegro/a",
"Hermano/a",
"Cuñado/a",
"Sobrino/a",
"Abuelo/a",
"Otro familiar",
"Otro no familiar"))
eder_personas <- eder_personas_br %>% left_join(cod_rel_par, by = "p4")Joins!!!
dplyr tiene las funciones típicas de join para el pareo de data.frames dependiendo la relación que deseemos. Ver más aquí.
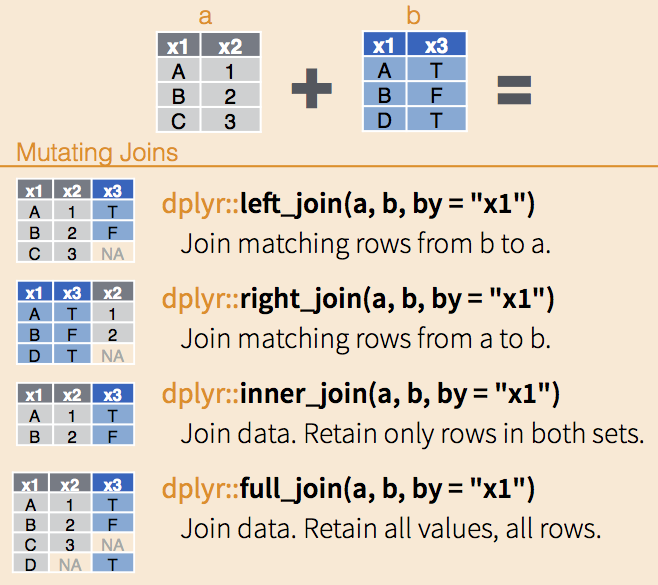
¿Los valores de p4_descripc coinciden con las categorías p4? Veamos el resultado creando una tabla de contingencia entre códigos y etiquetas: podemos usar la función count para descubrirlo: eder_personas %>% count(p4, p4_descripc). En caso de ver algún error, intentemos corregirlo:
eder_personas %>% count(p4, p4_descripc)
cod_rel_par <- data.frame(p4 = 1:14,
p4_descripc = c("Jefe/a",
"Cónyuge/pareja",
"Hijo/a",
"Hijastro/a",
"Yerno o nuera",
"Nieto/a",
"Padre/madre/suegro/a",
"Hermano/a",
"Cuñado/a",
"Sobrino/a",
"Abuelo/a",
"Otro familiar",
NA,
"Otro no familiar"))
eder_personas <- eder_personas_br %>% left_join(cod_rel_par, by = "p4")
eder_personas %>% count(p4, p4_descripc)Ahora sí, obtegamos la edad promedio de las personas encuestadas según relación de parentesco:
Parece una tabla grande (¿cuántas filas tiene?). Si querés mirar la tabla: View(tabla).
3.2.4 transformar (mutate), filtrar (filter), ordenar (arrange)
Mediante mutate podemos crear variables nuevas que pueden o no relacionarse a las existentes. Por ejemplo, hacemos una variable que nos diga si la persona es mayor de edad es_mayor, utilizando la función ifelse, la opción base de R.
# a que base nos referimos
head(eder_personas)
# creo una variable (sin resumir como en summarise)
eder_mayores <- eder_personas %>% mutate(es_mayor = ifelse(p3b >= 18, 1, 0))
# Nos avisan desde IDECBA que hay una hipótesis a corroborar de subdeclaración de edad por un 2%. Tenemos que corregir la edad y volver a armar la variable:
eder_mayores <- eder_personas %>% mutate(edad_altern = p3b * 1.02,
es_mayor = ifelse(p3b >= 18, 1, 0)) Podemos ordenar la tabla para ordenar de mayor a menor con arrange:
eder_mayores <- eder_mayores %>% arrange(edad_altern)
eder_mayores
# mejor al revés: descendente
eder_mayores <- eder_mayores %>% arrange(desc(edad_altern))¡OJO! ¿Qué son esas edades? Si vamos al diccionario, vemos que p3b = 9999 corresponde a No sabe o no responde. Entonces, nos conviene filtrarlos, utilizando filter. Aunque primero chequeemos el rango de edades, ¿no? Y nos aseguramos de que el filtro se haya hecho bien
table(eder_mayores$p3b) # La edad coherente más alta es 103
eder_mayores <- eder_mayores %>% filter(p3b <= 103) # Operador lógico! ¿te acordás?
range(eder_mayores$p3b)Vayamos hacia algo un poquito más complejo. Quiero obtener edad promedio de las personas que hicieron la encuesta retrospectiva por máximo nivel educativo alcanzado, pero solo si fueron lxs jefxs o cónyuges.
Comencemos por crear el data.frame con ese listado:
# busco un listado que cumpla las condiciones:
selec_jef_cony <- eder_mayores %>%
filter(retro == 1, # Seleccionados para la encuesta retrospectiva
p4 %in% 1:2) %>% # p4 sea Jefx o cónyuge
group_by(e_nivel) %>%
summarise(edad_media = mean(p3b),
edad_media_alt = mean(edad_altern)) ¿Y si quisiera hacer lo mismo, pero diferenciando por sexo? Y tomamos solo la edad que calculamos como alternativa
selec_jefcony_sex <- eder_mayores %>%
filter(retro == 1, # Seleccionados para la encuesta retrospectiva
p4 %in% 1:2) %>% # p4 sea Jefx o cónyuge
group_by(p2, e_nivel) %>%
summarise(edad_media = mean(p3b),
edad_media_alt = mean(edad_altern)) |>
select(p2, e_nivel, edad_media_alt)Si tuviéramos que relatar lo que estamos haciendo, sería algo del tipo: “tomá la eder, filtrame para traer solo jefxs y cónyuges que hayan contestado la retrospectiva, agrupámelos por estas variables y calculá el indicador para cada grupo; finalmente selecciona qué quieres mostrar”. Es una ¡composición de funciones!.
Otra operación común es la de calcular distribuciones en una variable, segmentando con determinada agrupación. Podemos ver el porcentaje de mujeres que contestó el cuestionario retrospectivo por año de nacimiento. Considerar que cualquier operación resumen (media, suma, contar) en un data.frame agrupado resumirá por grupos. Aprovechemos eso:
Porc_mujeres <- eder_mayores |>
filter(retro == 1) |>
group_by(p3, p2) |>
summarise(N = n()) |>
mutate(porc = N/sum(N) *100) |>
filter(p2 == 2) |>
select(p3, porc)Pero esto no es tan útil. Entonces podemos hacer esa distribución por cohorte de nacimiento agrupada, recodificando el año de nacimiento con la función case_when en una nueva variable.
unique(Porc_mujeres$p3)
distr_coh <- eder_mayores %>%
filter(retro == 1) |>
mutate(coh = case_when(
p3 %in% 1948:1952 ~ "1948-1952",
p3 %in% 1968:1972 ~ "1968-1972",
TRUE ~ "1978-1982"
)) %>%
group_by(coh, p2) |>
summarise(N = n()) |>
mutate(porc = N/sum(N) *100) |>
filter(p2 == 2) |>
select(coh, porc)3.2.6 Actividad
Crea un nuevo objeto con el nombre que desees a partir de eder_personas_br renombrando las variables a tu gusto para que los nombres guarden relación con su contenido.
Ordénala de la siguiente manera: ascendente ID de vivienda, ascendente en n° de hogar, y decreciente en la edad.
La vivienda identificada como 1001 no debería ser incluida en la base. Remover utilizando
filter.Podrías encontrar las viviendas (sus ID) que poseen más de un hogar? ¿Cuántas son y qué porcentaje representa del total de viviendas?
Crear una variable codificada para mayor de edad de carácter numérico (1 = mayor, 0 = menor) y su descripción (“Mayor de edad”, “Menor de edad”)
Filtra por elegibilidad para contestar la encuesta (Nota: recordar que para ser elegible para la encuesta la persona debía haber nacido en las cohortes 1948-1952, 1968-1972 o 1978-1982), y mostrar el total de individuos (utilizando
n()) por cohorteMediante la función mean obtén el promedio de edad de las personas que contestaron la encuesta por relación de parentesco con el jefe de hogar
Realiza lo anterior (filtrar y agrupar por cohorte y obtener el promedio de edad por relación de parentesco) en una sola sentencia. El resultado final debería mostrar cohorte, relación de parentesco y media de edad.
En una sola sentencia obtené la edad mediana por nivel educativo de las personas que contestaron la encuesta (utilizando
filter(retro == 1))Identifica (toma el ID de) un hogar cualquiera con 5 miembros.
3.2.7 Recursos adicionales
r4ds. Una biblia que debés llevar bajo el brazo.
dplyr: web del paquete.
Hoja de ayuda (o “cheat sheet”).
